こんにちは!ぼりたそです!
今回は次元削減法の一つであるUMAP(Uniform Manifold Approximation and Projection)についてわかりやすく解説した記事を作成しました。
この記事は以下の要点でまとめています。
- UMAPとは?
- なぜUMAPが必要なのか
- UMAPのアルゴリズム
- Pythonで実装
他の次元削減圧縮法としてPCAやt-SNEについて解説した記事もあるので、興味がある方は参照いただければと思います。
UMAPとは?
UMAP(Uniform Manifold Approximation and Projection)は、次元削減の手法の一つであり、高次元データを低次元に変換して視覚化や解析を容易にする技術です。特に、類似データがグループとしてまとまりやすいように非線形な構造を保持しながら、高速に処理できる点が特徴です。
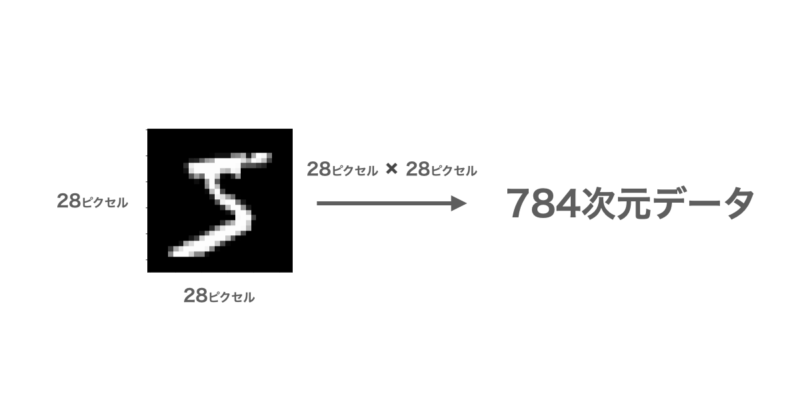
例えば、手書き数字データ(MNIST)は下図のように28×28ビットの合計784ビットとして表され、各ビットは白から黒までの濃淡を表す0~255の数値が格納されています。

このMNISTが0~9の数字について10000個ある場合、UMAPを実行すると次のように二次元グラフでそれぞれの数字ごとにクラスターとして可視化することができます。
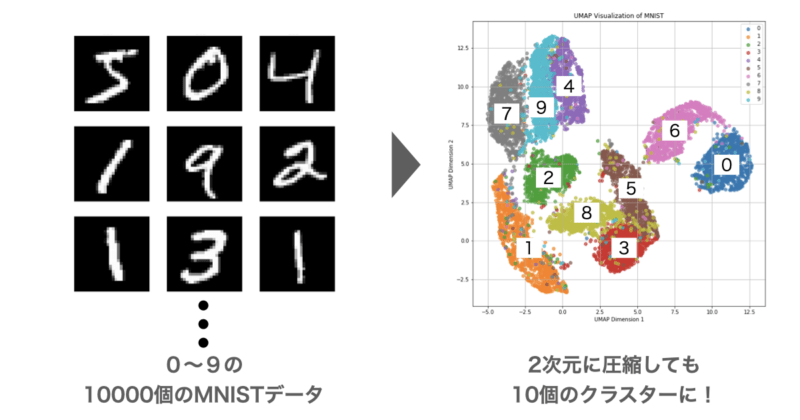

このようにUMAPでは高次元データを類似データがクラスターとしてまとめるように低次元データへと圧縮することができます。
なぜUMAPが必要なのか?
次になぜUMAPが必要なのかを他の次元圧縮手法であるPCA、t-SNEと比較しつつ解説していきます。
下の表に簡単ですが各手法の強み、弱み、使用場面についてまとめてみました。
| 手法 | 強み | 弱み | 使用場面 |
|---|---|---|---|
| PCA | ・線形情報の保持 ・計算が速い | ・非線形情報が捉えられない ・クラスタが重なりがち | ・前処理 ・特徴量選択 ・全体構造の把握 |
| t-SNE | ・局所構造の保持 ・クラスタの明確な分離 | ・計算が遅い ・大域構造が歪む | ・クラスタの可視化 ・類似データの探索 |
| UMAP | ・局所/大域構造のバランス ・高速な計算 | ・ハイパーパラメータ調整が難しい | ・大規模データの可視化 ・クラスタリングの補助 |
どの手法も場面によって向き不向きがあるのですが、敢えてUMAPとの比較を行なっていきます。
まず、PCAは計算が早く、線形情報の保持ができる手法でありますが、非線形情報を捉えることができず、次元削減後にクラスターが重なりがちになります。
この点、UMAPは非線形情報を捉えつつ、次元削減後にクラスターも分けることができ、PCAより優れている点といえます。
次にt-SNEは高次元の局所構造を保持することで次元削減後にクラスターごとにきちんと分けることができますが、データ数が多いと計算が遅く、大域構造(クラスター間の構造)がきちんと捉えられない場合が多いです。
この点、UMAPは局所構造と大域構造のバランス良く次元削減可能であり、計算も高速であるため、t-SNEよりも優れている点といえます。
一方でUMAPにもハイパーパラメータの調整が難しいという弱点がありますが、ベイズ最適化やグリッドサーチなど、自動でパラメータを決定できる手法がありますので、あまり大きな弱点というわけではありません。
以上からUMAPはPCAやt-SNEでは不向きな大規模データ可視化およびクラスタリングの可視化に必要不可欠な次元圧縮法と言えます。
UMAPのアルゴリズム
次にUMAPのアルゴリズムについて説明していきます。
UMAPの目的は、高次元データを低次元に圧縮しながら、データの構造や関係性をできるだけ保つことです。
これにより、視覚的にデータの分布を理解しやすくしたり、クラスタ構造を発見したりできます。
これを実現するために以下のステップで次元圧縮を行います。
- 高次元における確率近傍モデルの構築
- 低次元における確率モデルの構築
- コスト関数の定義
- コスト関数の最適化
以下順に説明していきます。
高次元における確率近傍モデルの構築
まずは高次元における確率近傍モデルの構築について説明していきます。
UMAPは、高次元空間でのデータ点同士の類似性を表すために 近傍グラフ(k-NN グラフ) を構築します。具体的には、データ点 $x_i$ の近傍点集合を考え、確率的に重み付けされたグラフ を作成します。
実際には以下の式で定義されます。
$$p_{ij} = \exp\left( \frac{-d(x_i, x_j) – \rho_i}{\sigma_i} \right)$$
$d(xi,xj)$ は $x_i$ と $x_j$ の距離。通常はユークリッド距離を使用。
$\rho_i$ はn_neighbors 個目の「最も近い点との距離」
$\sigma_i$ はスケールパラメータ(適応的に決定)
n_neighbors はハイパーパラメータであり、この値が大きければデータがより大局的構造を強調し、小さければ局所構造(クラスター)を強調する傾向があります。
また、上の条件付き確率を対称化した確率 $P_{ij}$ を定義し、これを高次元空間におけるデータの近さとします。
$$P_{ij} = p_{ij} + p_{ji} – p_{ij} p_{ji}$$
このとき、対称化するのは点 $x_i$ から見た $x_j$ の近さと、点 $x_j$ から見た $x_i$ の近さを平均化したいからです。
$p_{i|j}$ と $p_{j|i}$ は一般に異なっており、
例えば、点 $x_i$ の周りにたくさんの点があり、点$x_j$の周りにはあまり点がない場合、$p_{j|i}$ は小さくなるが$p_{i|j}$ は大きくなってしまいます。そうなると近さの扱いが煩雑になるため対称化を行なっています。
低次元における確率モデルの構築
次に低次元空間でも、高次元で得られた近傍関係を再現するような確率分布を構築していきます。
具体的にUMAPでは、双曲線相関関数を用いて、低次元空間での確率を以下の式で定義する。
$$q_{ij} = \left( 1 + a d(y_i, y_j)^{2b} \right)^{-1}$$
$d(yi_, y_j)$ は低次元空間におけるデータ点の距離関数。通常はユークリッド距離を使用。
$a, b$ はデータセットに応じて調整されるハイパーパラメータ
ちなみに$d(y_i, y_j)$が0にならないように最小距離(min_dist)を設定する必要があります。
これにより最適化時に距離がmin_dist以下にならないように調整されるため、データの局所構造を適度に維持しながらも、データの過度な密集を防ぎます。
コスト関数の定義
次にコスト関数の定義を行います。
具体的にUMAPはコスト関数をクロスエントロピー損失として定義し、これを最小化することで高次元と低次元のデータ距離がなるべく一致するようにしています。
$$\mathcal{L} = \sum_{i \neq j} P_{ij} \log q_{ij} + (1 – P_{ij}) \log (1 – q_{ij})$$
式の説明として、
第1項:高次元で近い点($P_{ij}$ が大きい点)同士が、低次元でも近くなるようにする。
第2項:高次元で遠い点($P_{ij}$ が小さい点)同士が、低次元でも遠くなるようにする。
このコスト関数の最小化を目指していきます。
コスト関数の最適化
最後にコスト関数を最小化します。
具体的には確率的勾配降下法という手法を用いて最適化します。
確率的勾配降下法とは簡単に言えば、「コスト関数の値を小さくする方向へ少しずつパラメータを更新する」 方法で以下の式に従って更新します。
$$y_i^{(t+1)} = y_i^{(t)} – \eta \frac{\partial \mathcal{L}}{\partial y_i}$$
また、$\eta$ は学習率であり勾配に対してどれだけ進むかを表しています。大きすぎると最小値を過ぎてしまい解が収束せず、小さすぎると最小化まで膨大な時間がかかってしまいます。
t-SNEでもコスト関数を最適化する際に似た方法で勾配降下法を用いていますが、確率的勾配降下法はより高速な計算を可能としています。その分、精度はやや低いといった側面もあります。
以上のアルゴリズムから高次元データを低次元に圧縮しながら、データの構造や関係性をできるだけ保つことができます。
Pythonで実装
それでは実際にPythonを使用してUMAPを実行してみましょう。
今回は冒頭で紹介した0〜9の手書き数字(MNIS)のデータ10000件をt-SNEにより二次元グラフに落とし込みます。
実行したコードは以下の通りです。
import numpy as np
import matplotlib.pyplot as plt
import umap
from sklearn.datasets import fetch_openml
# MNIST データセットのダウンロード
mnist = fetch_openml('mnist_784', version=1, as_frame=False)
X = mnist.data
y = mnist.target.astype(int)
# 計算量削減のため、ランダムに 10000 件を抽出
np.random.seed(42)
indices = np.random.choice(X.shape[0], 10000, replace=False)
X_subset = X[indices]
y_subset = y[indices]
# UMAP モデルの作成と適用
umap_model = umap.UMAP(n_components=2, n_neighbors=10, min_dist=0.3, random_state=42)
X_umap = umap_model.fit_transform(X_subset)
# プロットの設定
plt.figure(figsize=(10, 10))
# 各クラス(0〜9)のプロット
for i in range(10):
indices = y_subset == i
plt.scatter(X_umap[indices, 0], X_umap[indices, 1], label=str(i), alpha=0.7)
# プロットの装飾
plt.legend()
plt.title("UMAP Visualization of MNIST")
plt.xlabel("UMAP Dimension 1")
plt.ylabel("UMAP Dimension 2")
plt.grid(True)
plt.savefig('UMAP.png')
plt.show()
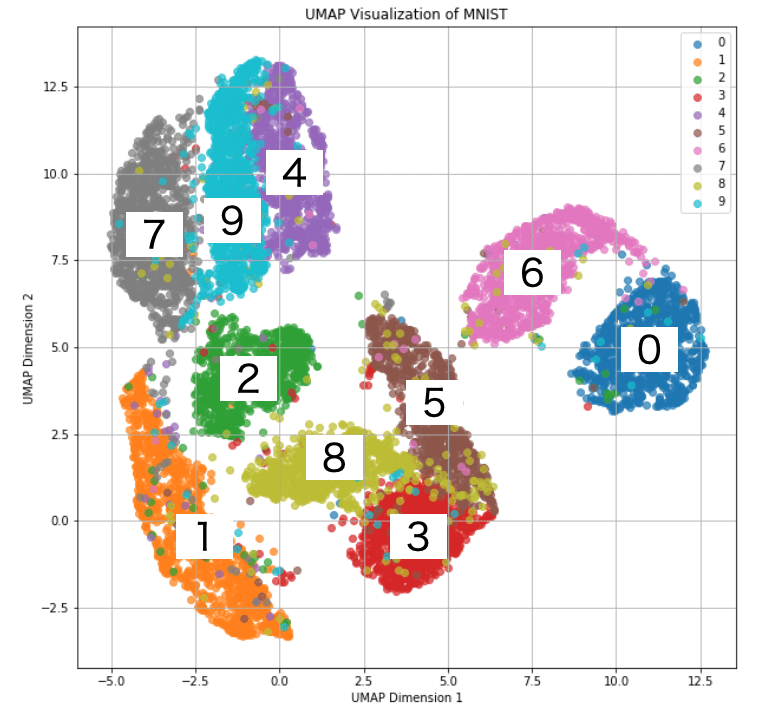
実行結果は以下のグラフのようになっており、元々784次元だったデータを二次元に落とし込んでも0~9の数字ごとにきちんと10個のクラスターに分かれていることがわかります。
また、似たような数字の形をしている「4」、「7」、「9」や「3」、「8」、「5」はクラスター同士が近い傾向が読み取れます。

以上がPythonによるUMAPの実行方法になります。
終わりに
以上がUMAPについての解説になります。UMAPは計算速度が高速でデータの非線形性の関係も捉えつつ低次元に圧縮することができるため、近年では主流の次元圧縮法となっています。機械学習のデータ前処理だけでなく、クラスタリングの可視化手法としても使用できるので、皆さんもぜひお試しいただければと思います。