こんにちは!ぼりたそです!今回はPythonのライブラリであるRDKitを使用して化合物の構造式を描画してみました。
この記事は以下のポイントでまとめています。
- RDkitとは?
- RDkitのインストール
- 化合物の可視化
- オススメの書籍
以前、構造式をテキスト表記するSMILESについてご紹介しましたが、今回はSMILESやその他の記述子から構造式を描画する方法について解説していきます。
RDkitとは?
まず、RDKitが何者かについてご紹介します。RDKitとはPythonのライブラリの一つであり、化合物の情報を取り扱うことのできるオープンソースライブラリとなっています。
RDKitの機能としては主に以下のようなものが挙げられます。
- 化学構造の読み込み
- 化合物情報の解析
- 化学反応のシミュレーション
- 化合物の可視化
全ての機能については解説しませんが、今回は化合物の可視化についてRDKitの機能を使っていきましょう!
RDkitについて詳細に知りたい方は以下URLよりドキュメントをご参照ください。
RDKitドキュメント↓↓↓
https://www.rdkit.org/docs/
RDKitのインストール
それでは早速、化合物の可視化を実行したいと思いますが、その前にもし RDKitをまだインストールしていない方は以下のコードを実行して RDKitをインストールしてください。
!pip instal RDKitちなみに上記のコードを実行してもインストールができない場合があるようです。おそらく、Pythonのバージョンが3.8以上であると RDKitが対応していないことが原因と思われます。
なので、もしインストールできなかった場合はPython3.7.9にダウングレードしてから RDKitをインストールしてみてください。環境を壊したくなければ仮想環境を作成して、Pythonをインストールしなおしてもいいかもしれません。
また、 RDKitを使用する際はjupyter notebookなどの画像や図が表示できる環境で実行するようにしましょう。
化合物の可視化
ではRDKitがインストールできた方はいよいよ化合物の可視化を実行してみましょう。
化合物を可視化するにはSMILESのような構造情報を読み込ませる必要があります。
今回は以下4つの構造情報をもとにそれぞれ化合物を可視化していきたいと思います。
- SMILES(Simplified Molecular Input Line Entry System)
- SMARTS(SMILES Arbitrary Target Specification)
- InChI(International Chemical Identifier)
- SDF(Structure Data File)
それでは順番に説明していきます。
SMILES
まずはSMILESを使用した化合物の可視化方法についてご紹介します。
SMILES(Simplified Molecular Input Line Entry System)は化合物の構造式をテキスト形式で表現するための表記法です。
構造式では人間からすれば視覚的に理解しやすいのですが、機械からしてみられば構造式からは何も理解できないのです。そこで、機械にもわかるように構造式を変換するのがSMILES式なのです。
詳しくは以下の記事にまとめてありますので、ご参照いただければと思います。
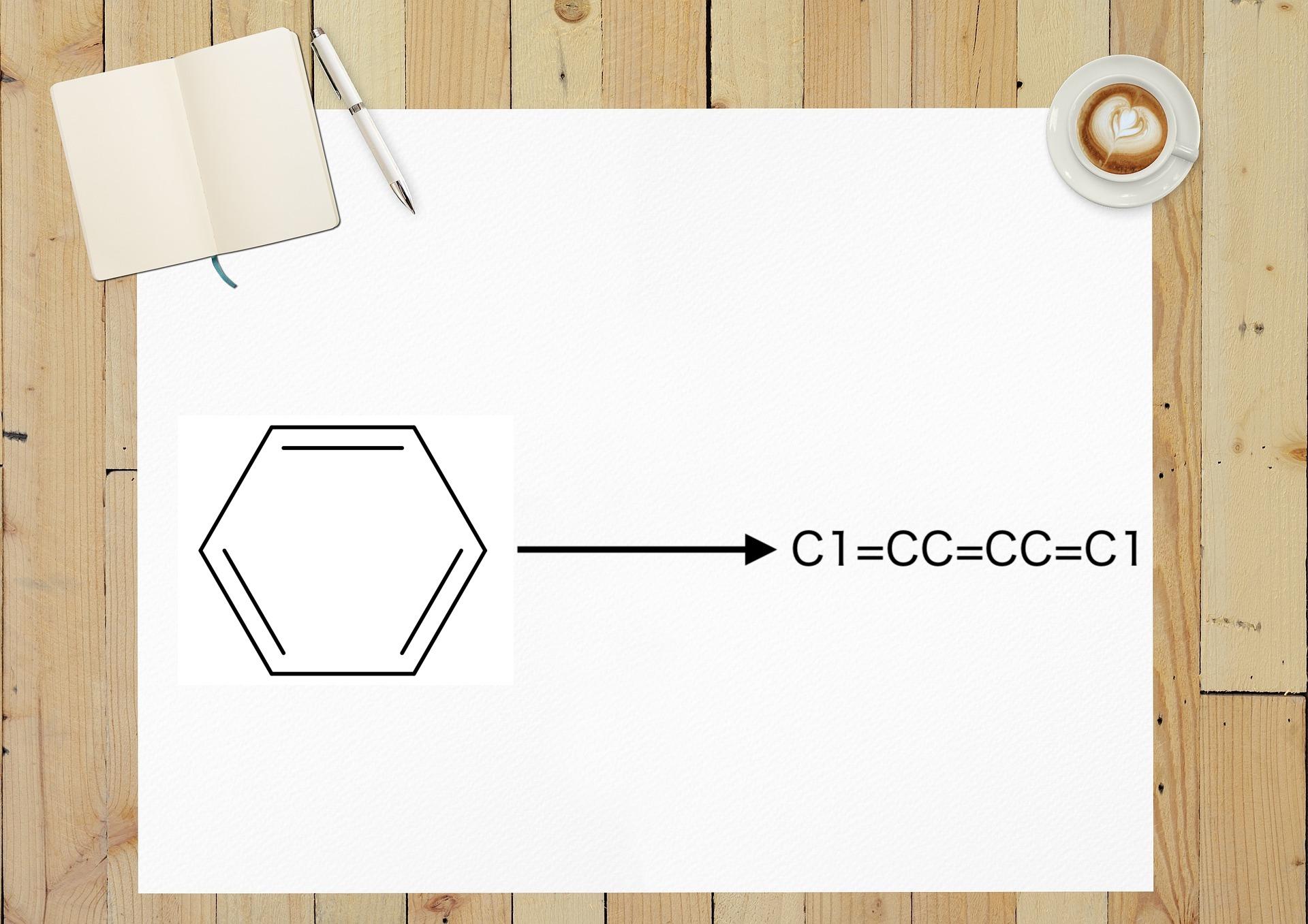
今回はベンゼンを可視化してみましょう。
ベンゼンの可視化のコードは以下の通りです。
from rdkit import Chem
from rdkit.Chem.Draw import IPythonConsole
# ベンゼンのSMILESを指定します
smiles = "c1ccccc1"
# SMILESをMolオブジェクトに変換します
mol = Chem.MolFromSmiles(smiles)
# ベンゼンを描画します
mol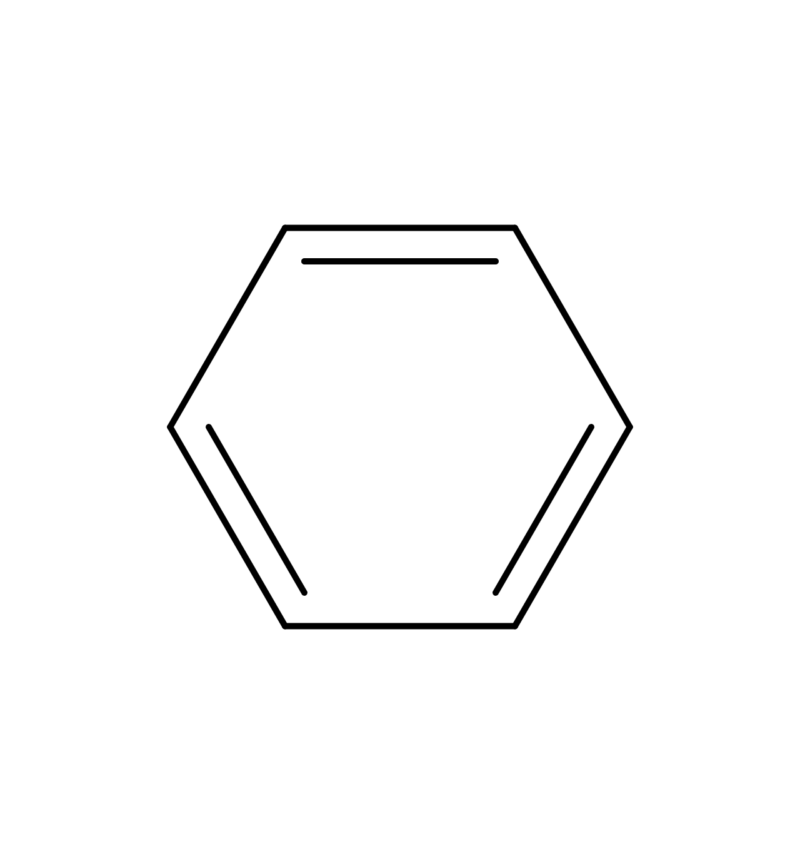

実際に実行してみると上の画像のようにベンゼンの構造式が描画されます。SMILESを読み込ませてChem.MolFromSmilesに引数として渡すだけなので簡単ですね。
ちなみに試しにアドレナリンも描画してみました。
from rdkit import Chem
from rdkit.Chem import Draw
# アドレナリンのSMILESを指定します
adrenaline_smiles = "CNCC(C1=CC(=C(C=C1)O)O)O"
# SMILESをMolオブジェクトに変換します
mol = Chem.MolFromSmiles(adrenaline_smiles)
# 描画結果を表示します
mol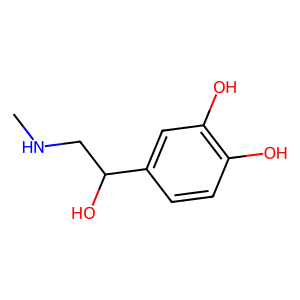

おお〜アドレナリンてこんな構造なんですねえ。適当にSMILESを引っ張ってきて描画してみましたが、案外簡単な形をしているんですね。生体ホルモンなのでもっと分子量が大きく複雑な構造をしていると思ってました。
また、RDKitでは2Dだけではなく3Dの描画も可能です。以下にベンゼンの分子を3Dで描画するコードを記載します。
from rdkit import Chem
from rdkit.Chem import AllChem
import py3Dmol
# SMILES 文字列
smiles = "c1ccccc1" # ベンゼン
# Molオブジェクトの生成と3D構造の計算
mol = Chem.MolFromSmiles(smiles)
mol = Chem.AddHs(mol)
AllChem.EmbedMolecule(mol)
AllChem.UFFOptimizeMolecule(mol)
# Molを3D描画
mb = Chem.MolToMolBlock(mol)
viewer = py3Dmol.view(width=300, height=300)
viewer.addModel(mb, 'mol')
viewer.setStyle({'stick': {}})
viewer.zoomTo()
viewer.show()
上記コードを実行すると以下のようにベンゼンの3D構造が描画されます。
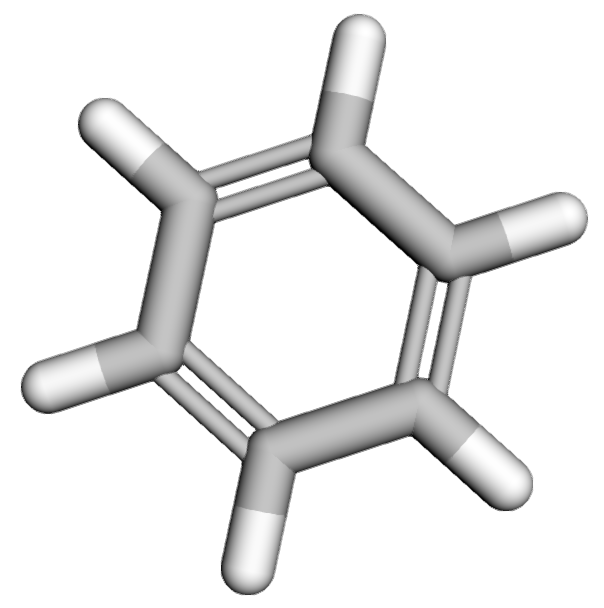

SMARTS
次にSMARTSを使用した化合物の可視化方法についてご紹介します。
SMARTSは、特定の部分構造を記述するためのSMILESの拡張バージョンです。
RDKitを使用するとSMARTSパターン(部分構造)から分子を検索し、描画することができます。
以下にSMARTSパターンとしてヒドロキシ基(ーOH)を設定し、エタノールの構造に含まれているヒドロキシ基を強調して可視化するコードを示します。
from rdkit import Chem
from rdkit.Chem import Draw
# SMARTSパターンを指定
smarts_pattern = "[OH]-[#6]" # 例: ヒドロキシ基
# SMARTSパターンに一致する分子を検索
mol = Chem.MolFromSmiles("CCO") # 例: エタノール
matches = mol.GetSubstructMatches(Chem.MolFromSmarts(smarts_pattern))
# 一致した部分を強調して描画
highlight_atoms = [atom for atom, _ in matches]
img = Draw.MolToImage(mol, highlightAtoms=highlight_atoms)
img.show()
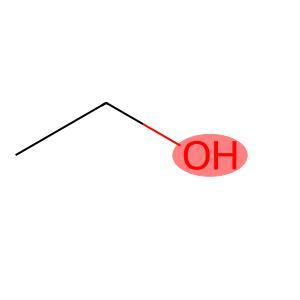

出力結果は上の通りでエタノールのヒドロキシ基部分が赤枠で強調されて可視化されていますね。
視覚的に一瞬で検索した部分構造がわかるのは便利ですね。
InChI
次にInChIを使用した化合物の可視化方法についてご紹介します。
InChIは化合物の国際的な識別子であり、SMILESとは異なる形式ですがSMILESと同様に化学構造をテキスト表記することができます。
InChIについては以下の記事にまとめてあるので参考いただければと思います。
今回はサリチル酸のInChIを使用して化合物を可視化してみましょう。コードは以下の通りです。
from rdkit.Chem import inchi
from rdkit.Chem import Draw
inchi_ = "InChI=1S/C7H6O3/c8-6-4-2-1-3-5(6)7(9)10/h1-4,8H,(H,9,10)" # 例: サリチル酸のInChI
mol = inchi.MolFromInchi(inchi_)
img = Draw.MolToImage(mol)
img.show()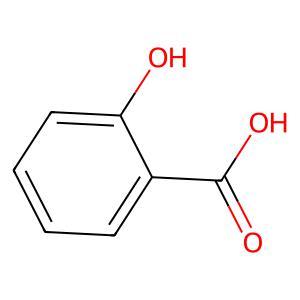

きちんとサリチル酸が描画されました。
SMILESと比較するとInChI表記の方がかなり長くなりますね。
IUPACと同じように国際規格の表記にするとルールが多くてどうしても長くなってしまうのでしょうか…
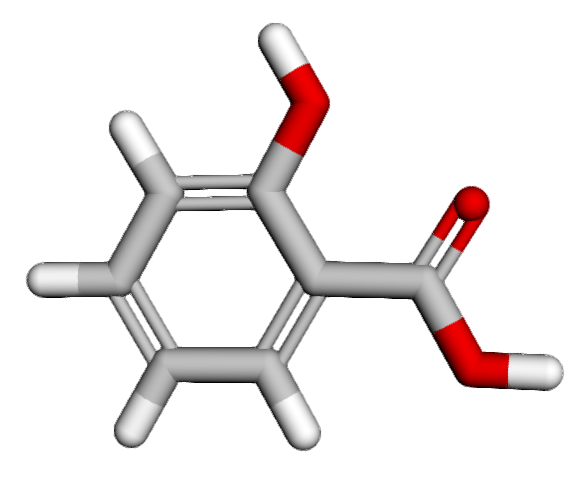
InChIについてもSMILESと同様に3D構造の描画も可能です。以下にPythonコードを記載します。
from rdkit.Chem import inchi
inchi_str = "InChI=1S/C7H6O3/c8-6-4-2-1-3-5(6)7(9)10/h1-4,8H,(H,9,10)" # 例: サリチル酸のInChI
mol = inchi.MolFromInchi(inchi_str)
mol = Chem.AddHs(mol)
AllChem.EmbedMolecule(mol)
AllChem.UFFOptimizeMolecule(mol)
mb = Chem.MolToMolBlock(mol)
viewer = py3Dmol.view(width=300, height=300)
viewer.addModel(mb, 'mol')
viewer.setStyle({'stick': {}})
viewer.zoomTo()
viewer.show()
上記コードを実行すると下のようにサリチル酸の3D構造が描画されます。

SDF
最後にSDFを使用して化合物を可視化する方法について紹介していきます。
SDFは、分子の3D構造とその他の情報を含むファイル形式です。RDKitはSDFファイルから分子を読み込み、描画することができます。
実際にベンゼンのSDFを使用して化合物を描画するコードを以下に示します。
from rdkit import Chem
from rdkit.Chem import AllChem
from rdkit.Chem import Draw
# SDFファイルから分子を読み込む
suppl = Chem.SDMolSupplier("benzene.sdf")#SDFファイルが保存してあるディレクトリを指定
mol = next(suppl)
# 分子を描画する
img = Draw.MolToImage(mol)
img.show()


実際にベンゼンが描画されましたね。
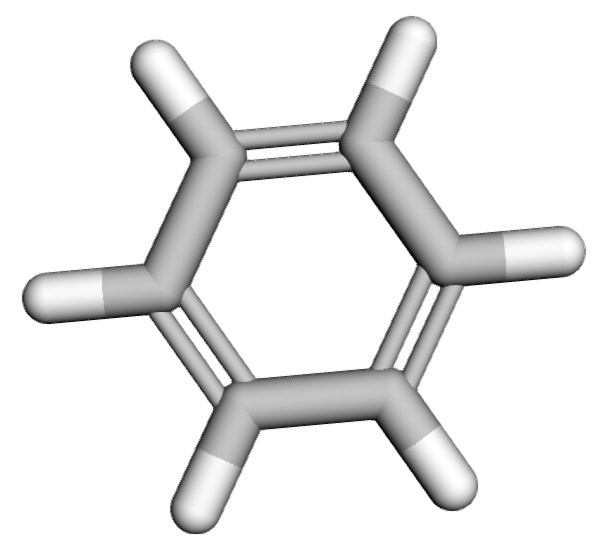
また、SDFファイルを用いて3D構造も描画することができます。以下にPythonの実行コードを記載します。
from rdkit import Chem
from rdkit.Chem import AllChem
import py3Dmol
# SDFファイルの読み込み(例:benzene.sdf)
supplier = Chem.SDMolSupplier("benzene_with_H.sdf")
mol = supplier[0]
# 水素原子を明示的に追加
mol_with_h = Chem.AddHs(mol, addCoords=True)
# 必要に応じて3D座標生成
if not mol_with_h.GetNumConformers():
AllChem.EmbedMolecule(mol_with_h)
AllChem.UFFOptimizeMolecule(mol_with_h)
# MolBlockとして書き出し(※ここが修正点)
mb = Chem.MolToMolBlock(mol_with_h)
# py3Dmolで3D可視化(stick 表示)
viewer = py3Dmol.view(width=400, height=400)
viewer.addModel(mb, 'mol')
viewer.setStyle({'stick': {}})
viewer.zoomTo()
viewer.show()
上記コードを実行すると以下のように描画されます。

ちなみにSDFファイルについてはPythonで生成することもできます。
参考程度に以下にコードを示します。
from rdkit import Chem
from rdkit.Chem import AllChem
# ベンゼン分子を作成
benzene = Chem.MolFromSmiles("c1ccccc1")
# 3D座標を生成
AllChem.EmbedMolecule(benzene, AllChem.ETKDG())
# SDFファイルを保存
sdf_file_path = "benzene.sdf" # 保存先のファイルパスを指定
sdf_writer = Chem.SDWriter(sdf_file_path)
sdf_writer.write(benzene)
sdf_writer.close()
print(f"ベンゼンのSDFファイルが {sdf_file_path} に保存されました。")
オススメの書籍
最後に、RDKitを使った化学構造の描画、そしてそれを応用した機械学習・データ分析に挑戦したい方に向けて、ぜひ手に取っていただきたい一冊をご紹介します。
ご紹介する『化学のためのPythonによるデータ解析・機械学習入門』では、SMILESをはじめとする化学構造のテキスト表記方法から、それらをPythonで数値化する方法まで、実践的なサンプルコード付きで丁寧に解説されています。
- SMILES記法の基本から応用まで丁寧に解説
- Pythonコード付きで即実践できる
- 化学構造のデータ化 → 機械学習まで一連の流れがわかる
- 実例豊富でイメージしやすい
- 数式少なめ、やさしい解説なので、初学者にも安心
上記の通り、本書籍の特徴として実際のデータセットを使ったデータ分析・機械学習の事例も豊富に紹介されており、単なる理論にとどまらず、研究や開発現場ですぐに応用できる実践力が身につきます。
難易度についても、タイトル通り入門書にふさわしく、高度な数学的知識は不要。大学の基礎レベルの知識があれば、スムーズに理解できる構成になっていますので、初めてこの分野に取り組む方でも安心です。
「SMILESを本当に使いこなせるようになりたい」、「化学×データサイエンスの第一歩を踏み出したい」という方に、強くおすすめできる一冊です!
終わりに
今回はPythonを使って様々な表記法から構造式を描画してみましたが、本格的で面白かったですね。
というかPythonで構造式を描画できるだなんて最初知った時は非常に驚いたのを覚えています。 Pythonさん、さすがっす…