こんにちはぼりたそです!
今回はPythonを使用して以前PubChemから取得したデータをもとにXlogPをベイズ最適化を実装してみました。
ベイズ最適化のコードは明治大学 金子弘昌先生が公開されているものを使用させていただいております。
金子弘昌先生がGithubにて公開されているコード
https://github.com/hkaneko1985/python_doe_kspub/blob/master/sample_program_05_04_bayesian_optimization.py
この記事は以下のポイントでまとめています。
- 最適化フロー
- 最適化条件
- 使用したプログラムコード
- ベイズ最適化結果
今回使用したデータを以下からダウンロードできます。data_yが目的変数、data_xが説明変数となっています。
※CSVではアクセスできないようなので、エクセルファイル(.xlsx)をダウンロードしてCSVとしてエクスポートしていただけますと使用できるかと思います。
それでは、今回実装したベイズ最適化の詳細についてご紹介していきます。
最適化フロー
まずは今回行った最適化フローを説明します。
フローについては以下のフローチャートの通りとなります。
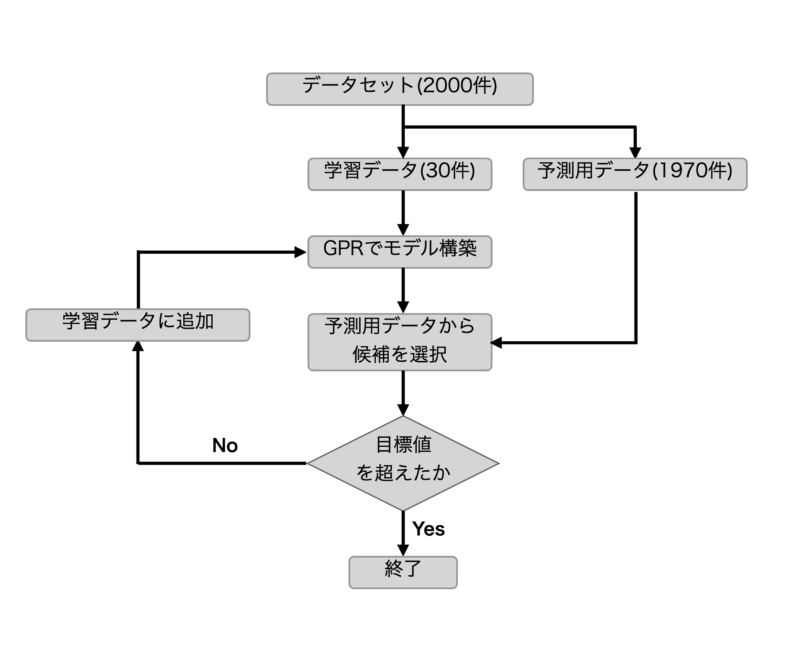

PubChemから取得したデータ約2000件を学習用データ(30件)と予測用データ1970件に分けます。
その後、学習用データからGPR(ガウス過程回帰)により学習モデルを構築し予測用データから次の候補を選択します。
候補が目標値を達成していれば探索終了とし、達成していなければ候補点を学習データに追加して再度モデル構築するフローとなっております。
最適化条件
続いてはベイズ最適化の条件について説明いたします。
主な条件は以下の通りです。
| 説明変数 | 化合物のMordred記述子 |
| 目的変数 | XlogP |
| カーネル | CVにより11種の中から選択 |
| 獲得関数 | EI(Expected Improvement) |
| 目標値 | 学習データの最大値の更新 |
説明変数は化合物のMordred記述子、目的変数はXlogPとなります。説明変数については特に次元削減などは行わずにそのまま使用しています。
カーネルは後程ご説明しますが、用意された11種の中からCVにより選択する形式となっています。
獲得関数はEIとなっており、学習データの更新値の期待値から次の候補を選択する獲得関数となっています。獲得関数については以下の記事にまとめていますので、ご参考にしていただければと思います。
目標値は学習データの最大値の更新としています。
使用したコード
次に今回のベイズ最適化に使用したコードをご紹介します。
データ分割
まずはデータセットを学習用データと予測用データに分割するコードです。
学習データ数を30個に設定し、データセットからランダムで抽出しています。抽出はランダムに行っていますが、再現性を担保するためにSeed値を設定しています。
#データセットを学習用データと予測用データに分割するコード
import pandas as pd
import random
# CSVファイルの読み込み
data_x = pd.read_csv('data_x.csv', index_col='cid')
data_y = pd.read_csv('data_y.csv', index_col='cid')
# 抽出する行数が元の行数より多い場合は、元の行数に合わせる
num_rows = min(30, len(data_x))
#Seed値を設定
seed = 0
# ランダムに行を抽出
random_rows = data_x.sample(n=num_rows, random_state=random.seed(seed))
# 抽出した行を別のファイルに保存する
random_rows.to_csv(f'train_x_seed_{seed}.csv', index='cid')
data_y.loc[random_rows.index].to_csv(f'train_y_seed_{seed}.csv', index='cid')
# 元のデータから抽出した行を削除
data_x = data_x.drop(random_rows.index)
data_y = data_y.drop(random_rows.index)
# 更新されたデータを別のファイルに保存する場合
data_x.to_csv(f'prediction_data_x_seed_{seed}.csv', index='cid')
data_y.to_csv(f'prediction_data_y_seed_{seed}.csv', index='cid')
ちなみに使用しているデータセットのXlogPのデータ分布を以下のコードから可視化してみました。
import pandas as pd
import matplotlib.pyplot as plt
# CSVファイルの読み込み
df = pd.read_csv('data_y.csv',index_col='cid')
# 1列目のデータを取得
column_data = df.iloc[:, 0]
# ヒストグラムの作成
plt.hist(column_data, bins=20, range=(-20,20)) # ビンの数は適宜調整してください
plt.xlabel('値')
plt.ylabel('出現回数')
plt.title('ヒストグラム')
# ヒストグラムの表示
plt.show()
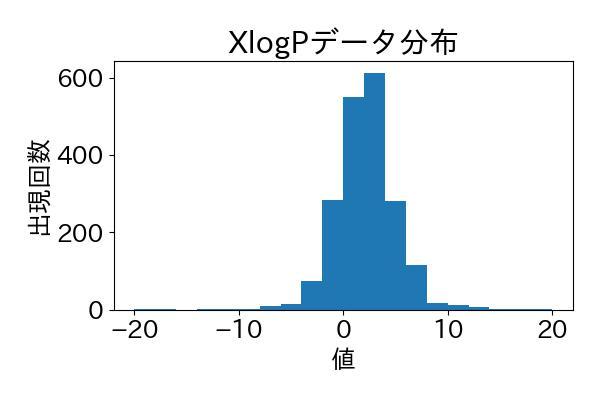

ランダムに取得したデータですが、XlogPの分布は綺麗に正規分布になっていますね。これならデータの偏りもなく、良好なモデルが構築できそうです。
ベイズ最適化
次にベイズ最適化に使用したプログラムですが、冒頭で記載の通り、明治大学 金子弘昌先生がGithubで公開されているコードを使用しています。
金子弘昌先生が公開しているコード↓↓↓↓↓
https://github.com/hkaneko1985/python_doe_kspub/blob/master/sample_program_05_04_bayesian_optimization.py
このコードはPythonを使用しており、学習データと予測用データをCSVファイルとして読み込めば実行できるようになっております。
その他にも以下のような特徴があります。
- カーネル:クロスバリデーションにより11種から選択 or 任意のカーネルを選択
- 獲得関数:PTR、PI、EI、MIから選択
今回はカーネルはクロスバリデーション(CV)より11種から選択し、獲得関数はEIを選択しています。
# -*- coding: utf-8 -*-
"""
@author: Hiromasa Kaneko
"""
import matplotlib.pyplot as plt
import pandas as pd
import numpy as np
from scipy.stats import norm
from sklearn.model_selection import KFold, cross_val_predict
from sklearn.gaussian_process import GaussianProcessRegressor
from sklearn.gaussian_process.kernels import WhiteKernel, RBF, ConstantKernel, Matern, DotProduct
from sklearn.metrics import r2_score, mean_squared_error, mean_absolute_error
regression_method = 'gpr_kernels' # gpr_one_kernel', 'gpr_kernels'
acquisition_function = 'EI' # 'PTR', 'PI', 'EI', 'MI'
fold_number = 10 # クロスバリデーションの fold 数
kernel_number = 2 # 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10
target_range = [0, 1] # PTR
relaxation = 0.01 # EI, PI
delta = 10 ** -6 # MI
dataset = pd.read_csv('data_x.csv', index_col=0, header=0)
x_prediction = pd.read_csv('remaining_data_x.csv', index_col=0, header=0)
# データ分割
y = dataset.iloc[:, 0] # 目的変数
x = dataset.iloc[:, 1:] # 説明変数
# 標準偏差が 0 の特徴量の削除
deleting_variables = x.columns[x.std() == 0]
x = x.drop(deleting_variables, axis=1)
x_prediction = x_prediction.drop(deleting_variables, axis=1)
# カーネル 11 種類
kernels = [ConstantKernel() * DotProduct() + WhiteKernel(),
ConstantKernel() * RBF() + WhiteKernel(),
ConstantKernel() * RBF() + WhiteKernel() + ConstantKernel() * DotProduct(),
ConstantKernel() * RBF(np.ones(x.shape[1])) + WhiteKernel(),
ConstantKernel() * RBF(np.ones(x.shape[1])) + WhiteKernel() + ConstantKernel() * DotProduct(),
ConstantKernel() * Matern(nu=1.5) + WhiteKernel(),
ConstantKernel() * Matern(nu=1.5) + WhiteKernel() + ConstantKernel() * DotProduct(),
ConstantKernel() * Matern(nu=0.5) + WhiteKernel(),
ConstantKernel() * Matern(nu=0.5) + WhiteKernel() + ConstantKernel() * DotProduct(),
ConstantKernel() * Matern(nu=2.5) + WhiteKernel(),
ConstantKernel() * Matern(nu=2.5) + WhiteKernel() + ConstantKernel() * DotProduct()]
# オートスケーリング
autoscaled_y = (y - y.mean()) / y.std()
autoscaled_x = (x - x.mean()) / x.std()
autoscaled_x_prediction = (x_prediction - x.mean()) / x.std()
# モデル構築
if regression_method == 'gpr_one_kernel':
selected_kernel = kernels[kernel_number]
model = GaussianProcessRegressor(alpha=0, kernel=selected_kernel)
elif regression_method == 'gpr_kernels':
# クロスバリデーションによるカーネル関数の最適化
cross_validation = KFold(n_splits=fold_number, random_state=9, shuffle=True) # クロスバリデーションの分割の設定
r2cvs = [] # 空の list。カーネル関数ごとに、クロスバリデーション後の r2 を入れていきます
for index, kernel in enumerate(kernels):
print(index + 1, '/', len(kernels))
model = GaussianProcessRegressor(alpha=0, kernel=kernel)
estimated_y_in_cv = np.ndarray.flatten(cross_val_predict(model, autoscaled_x, autoscaled_y, cv=cross_validation))
estimated_y_in_cv = estimated_y_in_cv * y.std(ddof=1) + y.mean()
r2cvs.append(r2_score(y, estimated_y_in_cv))
optimal_kernel_number = np.where(r2cvs == np.max(r2cvs))[0][0] # クロスバリデーション後の r2 が最も大きいカーネル関数の番号
optimal_kernel = kernels[optimal_kernel_number] # クロスバリデーション後の r2 が最も大きいカーネル関数
print('クロスバリデーションで選択されたカーネル関数の番号 :', optimal_kernel_number)
print('クロスバリデーションで選択されたカーネル関数 :', optimal_kernel)
# モデル構築
model = GaussianProcessRegressor(alpha=0, kernel=optimal_kernel) # GPR モデルの宣言
model.fit(autoscaled_x, autoscaled_y) # モデル構築
# トレーニングデータの推定
autoscaled_estimated_y, autoscaled_estimated_y_std = model.predict(autoscaled_x, return_std=True) # y の推定
estimated_y = autoscaled_estimated_y * y.std() + y.mean() # スケールをもとに戻す
estimated_y_std = autoscaled_estimated_y_std * y.std() # スケールをもとに戻す
estimated_y = pd.DataFrame(estimated_y, index=x.index, columns=['estimated_y'])
estimated_y_std = pd.DataFrame(estimated_y_std, index=x.index, columns=['std_of_estimated_y'])
# トレーニングデータの実測値 vs. 推定値のプロット
plt.rcParams['font.size'] = 18
plt.scatter(y, estimated_y.iloc[:, 0], c='blue') # 実測値 vs. 推定値プロット
y_max = max(y.max(), estimated_y.iloc[:, 0].max()) # 実測値の最大値と、推定値の最大値の中で、より大きい値を取得
y_min = min(y.min(), estimated_y.iloc[:, 0].min()) # 実測値の最小値と、推定値の最小値の中で、より小さい値を取得
plt.plot([y_min - 0.05 * (y_max - y_min), y_max + 0.05 * (y_max - y_min)],
[y_min - 0.05 * (y_max - y_min), y_max + 0.05 * (y_max - y_min)], 'k-') # 取得した最小値-5%から最大値+5%まで、対角線を作成
plt.ylim(y_min - 0.05 * (y_max - y_min), y_max + 0.05 * (y_max - y_min)) # y 軸の範囲の設定
plt.xlim(y_min - 0.05 * (y_max - y_min), y_max + 0.05 * (y_max - y_min)) # x 軸の範囲の設定
plt.xlabel('actual y') # x 軸の名前
plt.ylabel('estimated y') # y 軸の名前
plt.gca().set_aspect('equal', adjustable='box') # 図の形を正方形に
plt.show() # 以上の設定で描画
# トレーニングデータのr2, RMSE, MAE
print('r^2 for training data :', r2_score(y, estimated_y))
print('RMSE for training data :', mean_squared_error(y, estimated_y, squared=False))
print('MAE for training data :', mean_absolute_error(y, estimated_y))
# トレーニングデータの結果の保存
y_for_save = pd.DataFrame(y)
y_for_save.columns = ['actual_y']
y_error_train = y_for_save.iloc[:, 0] - estimated_y.iloc[:, 0]
y_error_train = pd.DataFrame(y_error_train)
y_error_train.columns = ['error_of_y(actual_y-estimated_y)']
results_train = pd.concat([y_for_save, estimated_y, y_error_train, estimated_y_std], axis=1) # 結合
results_train.to_csv('estimated_y_in_detail_{0}.csv'.format(regression_method)) # 推定値を csv ファイルに保存。同じ名前のファイルがあるときは上書きされますので注意してください
# クロスバリデーションによる y の値の推定
cross_validation = KFold(n_splits=fold_number, random_state=9, shuffle=True) # クロスバリデーションの分割の設定
autoscaled_estimated_y_in_cv = cross_val_predict(model, autoscaled_x, autoscaled_y, cv=cross_validation) # y の推定
estimated_y_in_cv = autoscaled_estimated_y_in_cv * y.std() + y.mean() # スケールをもとに戻す
estimated_y_in_cv = pd.DataFrame(estimated_y_in_cv, index=x.index, columns=['estimated_y'])
# クロスバリデーションにおける実測値 vs. 推定値のプロット
plt.rcParams['font.size'] = 18
plt.scatter(y, estimated_y_in_cv.iloc[:, 0], c='blue') # 実測値 vs. 推定値プロット
y_max = max(y.max(), estimated_y_in_cv.iloc[:, 0].max()) # 実測値の最大値と、推定値の最大値の中で、より大きい値を取得
y_min = min(y.min(), estimated_y_in_cv.iloc[:, 0].min()) # 実測値の最小値と、推定値の最小値の中で、より小さい値を取得
plt.plot([y_min - 0.05 * (y_max - y_min), y_max + 0.05 * (y_max - y_min)],
[y_min - 0.05 * (y_max - y_min), y_max + 0.05 * (y_max - y_min)], 'k-') # 取得した最小値-5%から最大値+5%まで、対角線を作成
plt.ylim(y_min - 0.05 * (y_max - y_min), y_max + 0.05 * (y_max - y_min)) # y 軸の範囲の設定
plt.xlim(y_min - 0.05 * (y_max - y_min), y_max + 0.05 * (y_max - y_min)) # x 軸の範囲の設定
plt.xlabel('actual y') # x 軸の名前
plt.ylabel('estimated y') # y 軸の名前
plt.gca().set_aspect('equal', adjustable='box') # 図の形を正方形に
plt.show() # 以上の設定で描画
# クロスバリデーションにおけるr2, RMSE, MAE
print('r^2 in cross-validation :', r2_score(y, estimated_y_in_cv))
print('RMSE in cross-validation :', mean_squared_error(y, estimated_y_in_cv, squared=False))
print('MAE in cross-validation :', mean_absolute_error(y, estimated_y_in_cv))
# クロスバリデーションの結果の保存
y_error_in_cv = y_for_save.iloc[:, 0] - estimated_y_in_cv.iloc[:, 0]
y_error_in_cv = pd.DataFrame(y_error_in_cv)
y_error_in_cv.columns = ['error_of_y(actual_y-estimated_y)']
results_in_cv = pd.concat([y_for_save, estimated_y_in_cv, y_error_in_cv], axis=1) # 結合
results_in_cv.to_csv('estimated_y_in_cv_in_detail_{0}.csv'.format(regression_method)) # 推定値を csv ファイルに保存。同じ名前のファイルがあるときは上書きされますので注意してください
# 予測
estimated_y_prediction, estimated_y_prediction_std = model.predict(autoscaled_x_prediction, return_std=True)
estimated_y_prediction = estimated_y_prediction * y.std() + y.mean()
estimated_y_prediction_std = estimated_y_prediction_std * y.std()
# 獲得関数の計算
cumulative_variance = np.zeros(x_prediction.shape[0]) # MI で必要な "ばらつき" を 0 で初期化
if acquisition_function == 'MI':
acquisition_function_prediction = estimated_y_prediction + np.log(2 / delta) ** 0.5 * (
(estimated_y_prediction_std ** 2 + cumulative_variance) ** 0.5 - cumulative_variance ** 0.5)
cumulative_variance = cumulative_variance + estimated_y_prediction_std ** 2
elif acquisition_function == 'EI':
acquisition_function_prediction = (estimated_y_prediction - max(y) - relaxation * y.std()) * \
norm.cdf((estimated_y_prediction - max(y) - relaxation * y.std()) /
estimated_y_prediction_std) + \
estimated_y_prediction_std * \
norm.pdf((estimated_y_prediction - max(y) - relaxation * y.std()) /
estimated_y_prediction_std)
elif acquisition_function == 'PI':
acquisition_function_prediction = norm.cdf(
(estimated_y_prediction - max(y) - relaxation * y.std()) / estimated_y_prediction_std)
elif acquisition_function == 'PTR':
acquisition_function_prediction = norm.cdf(target_range[1],
loc=estimated_y_prediction,
scale=estimated_y_prediction_std
) - norm.cdf(target_range[0],
loc=estimated_y_prediction,
scale=estimated_y_prediction_std)
acquisition_function_prediction[estimated_y_prediction_std <= 0] = 0
# 保存
estimated_y_prediction = pd.DataFrame(estimated_y_prediction, x_prediction.index, columns=['estimated_y'])
estimated_y_prediction.to_csv('estimated_y_prediction_{0}.csv'.format(regression_method)) # 予測結果を csv ファイルに保存。同じ名前のファイルがあるときは上書きされますので注意してください
estimated_y_prediction_std = pd.DataFrame(estimated_y_prediction_std, x_prediction.index, columns=['std_of_estimated_y'])
estimated_y_prediction_std.to_csv('estimated_y_prediction_{0}_std.csv'.format(regression_method)) # 予測値の標準偏差を csv ファイルに保存。同じ名前のファイルがあるときは上書きされますので注意してください
acquisition_function_prediction = pd.DataFrame(acquisition_function_prediction, index=x_prediction.index, columns=['acquisition_function'])
acquisition_function_prediction.to_csv('acquisition_function_prediction_{0}_{1}.csv'.format(regression_method, acquisition_function)) # 獲得関数を csv ファイルに保存。同じ名前のファイルがあるときは上書きされますので注意してください
# 次のサンプル
next_sample = x_prediction.loc[acquisition_function_prediction.idxmax()] # 次のサンプル
next_sample.to_csv('next_sample_bo_{0}_{1}.csv'.format(regression_method, acquisition_function)) # csv ファイルに保存。同じ名前のファイルがあるときは上書きされますので注意してくださいベイズ最適化の結果
先ほど紹介したコードから実際にmordred記述子を説明変数、XlogPを目的関数としてベイズ最適化を実行してみました。最適化の細かい条件は以下の通りです。
- 説明変数:Mordred記述子
- 目的変数:XlogP
- カーネル:11種の中からCVにより選択
- 獲得関数:EI(Expected Improvement)
- 目標値:予測データの最大値(XlogP=22.4)
今回は学習データを10通り用意してそれぞれ最適化を実行してみました。
| バッチ | 実探索数 |
|---|---|
| 1 | 6 |
| 2 | 4 |
| 3 | 26 |
| 4 | 9 |
| 5 | 5 |
| 6 | 7 |
| 7 | 4 |
| 8 | 5 |
| 9 | 10 |
| 10 | 14 |
| 平均 | 9 |
表の通り、10バッチの学習データにてベイズ最適化を実行し、それぞれの目標値を達成するまでの実探索数を記載しました。
平均探索数は9回とかなり高い少ない探索数で目標値に達していることがわかります。今回は特に主成分分析をしていないのですが、こんなに精度良く出るものなんですね…
今回は構造からXLogPを予測しているので、ある程度予測しやすいモデルだったこともあるのでしょうが、それにしても予測精度が良いという印象を受けました。
終わりに
いかがでしたでしょうか!今回はPubChemから取得した化合物データを使用してベイズ最適化をしてみました。精度としては良すぎて怖いくらいでしたね。個人的には説明変数の次元を削減したり、獲得関数によってどの程度探索数が変化するのか検証してみたいですね。





