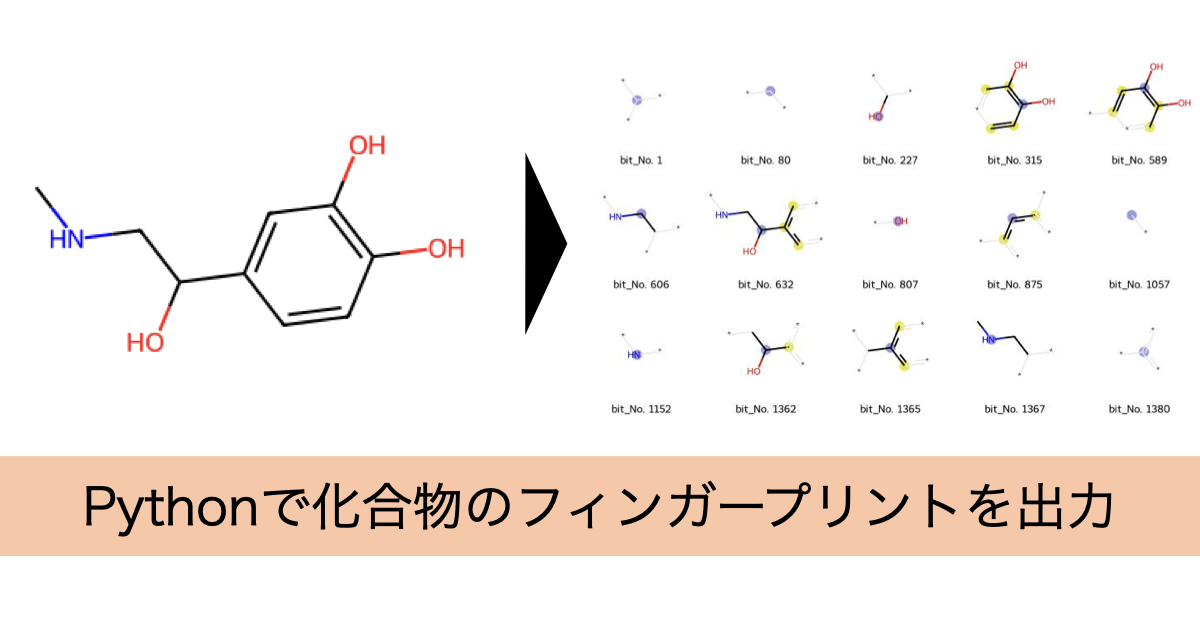
こんにちは!ぼりたそです!
今回はフィンガープリントの部分構造を可視化する方法について解説した記事を作成してみました。
- フィンガープリントとは?
- 部分構造の可視化
- 共通する部分構造の可視化
それでは詳細に解説していきます。
フィンガープリントとは
まず化学構造におけるフィンガープリントについて説明していきます。
フィンガープリントは、分子の構造や性質をバイナリビット(0または1)や数値の形式で表現するものであり、分子構造の特定のパターンや特性を分析することができます。
主な使われ方としては、類似性評価や分子の検索、クラスタリング、機械学習などが挙げられ、分子同士の構造的な比較に役立ちます。
例えば下の図のようにアドレナリンの構造があったとします。
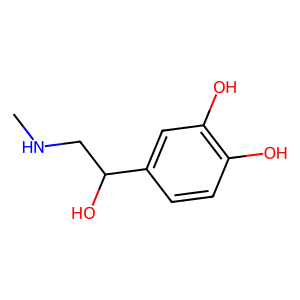
フィンガープリントに変換した場合、ベンゼン環の部分構造を含むか?というビットに対しては含むので「1」となりますが、カルボキシル基の部分構造を含むか?というビットに対しては含まないので「0」と記述されます。
このように部分構造を含むかどうかを「0」か「1」で表し、それを数百個並べることで分子構造の特徴を記述しているわけですね。
部分構造の可視化
では次に部分構造の可視化を行なっていきます。
今回はアドレナリンのMorganフィンガープリントを計算し、該当する部分構造を描画することにしました。
部分構造の可視化はPythonで行い、ライブラリはrdkitを使用しています。
コードは以下の通りで、まず分子のSMILESを定義して、Molオブジェクトを生成します。
次にMorganフィンガープリントを計算しますが、この際に中心原子からの半径と「1」であるビット情報を格納する空の辞書を定義しています。
最後にビットが1である部分構造を描画していますが、ここではlist_bits_infoとして化合物のMolオブジェクト、ビットNo.、ビット情報(0 or 1)を一つのタプルとして格納しています。
ビット情報が格納されたタプルをDrawMorganBitsメソッドに引数として渡すことでビットが1である部分構造を描画しています。
from rdkit import Chem
from rdkit.Chem import AllChem
from rdkit.Chem import Draw
# 分子のSMILES表記を指定
molecule_smiles = "CNCC(C1=CC(=C(C=C1)O)CO)O" # ここに適切な分子のSMILES表記を入力してください
# SMILESからMolオブジェクトを生成
molecule = Chem.MolFromSmiles(molecule_smiles)
# Morganフィンガープリントを計算
radius = 2 # Morganフィンガープリントの半径を指定
bit_info = {} #bitが1である情報を入れる空の辞書
mfp = AllChem.GetMorganFingerprintAsBitVect(molecule, radius, bitInfo=bit_info)
# ビットが1である部分構造を描画
list_bits_info = ((molecule, bit, bit_info) for bit in bit_info.keys())
legend = [f'bit_No. {str(i)}' for i, bit in enumerate(mfp) if bit == 1]
Draw.DrawMorganBits(list_bits_info, molsPerRow=5, legends=legend)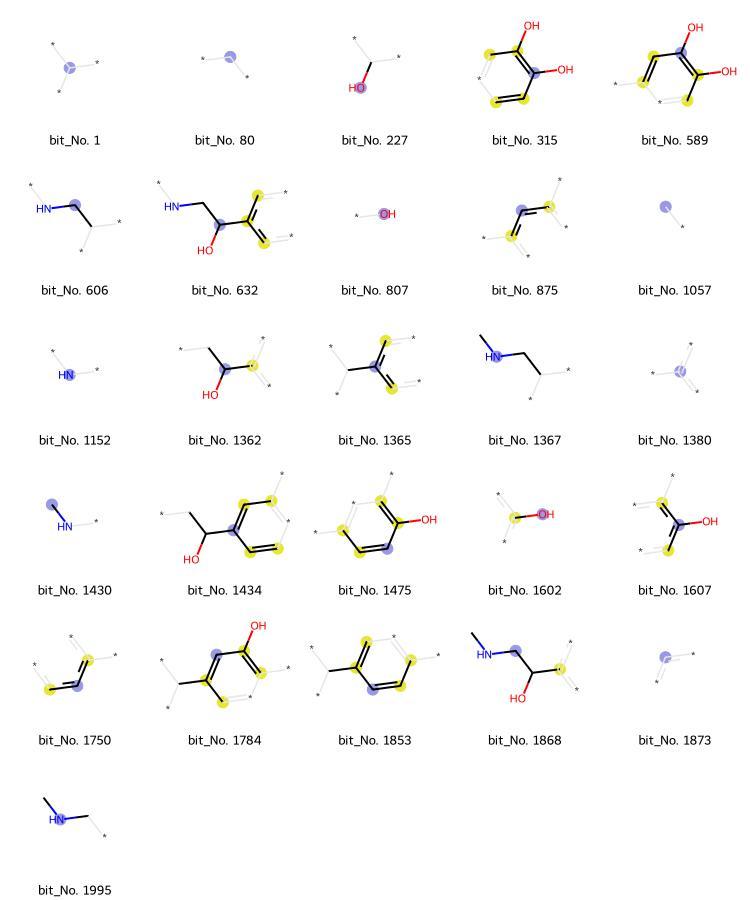
描画されたのは全部で31個でした。
青い丸で表示されているのが書くビットにおける中心原子になります。中心原子はこちらから指定することもでき、反応活性に注目したい場合は反応点の原子を中心原子とするのが良いとされています。
ちなみにmolPerRowの引数を変更すれば一行当たりに描画する部分構造の数を調整することができます。
共通する部分構造の可視化
それでは次に2つの化合物に共通する部分構造を可視化してみました。
今回はアドレナリンとドーパミンの共通する部分構造を可視化することにしました。
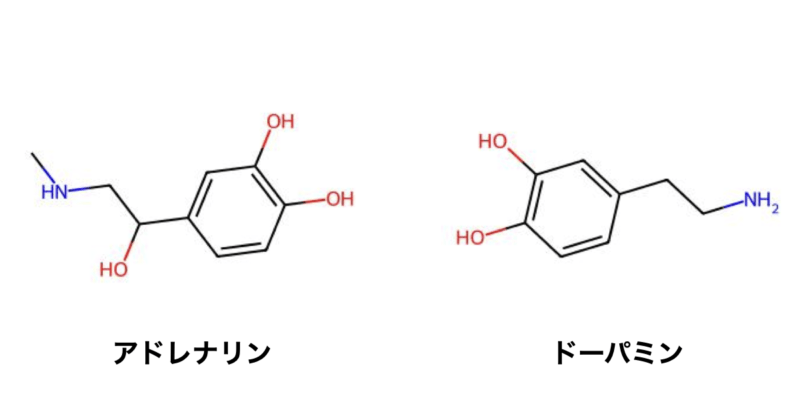
可視化方法は先ほどと同じようにPythonのrdkitライブラリを使用しており、Morganフィンガープリンで可視化するといったものです。
from rdkit import Chem
from rdkit.Chem import AllChem
from rdkit.Chem import Draw
#SMILESのリストを作成
smiles_list = ["CNCC(C1=CC(=C(C=C1)O)O)O", "c1cc(c(cc1CCN)O)O"]
# SMILESからMolオブジェクトを生成
for smiles in smiles_list:
com_1 =
adrenaline = Chem.MolFromSmiles(adrenaline_smiles)
dopamine = Chem.MolFromSmiles(dopamine_smiles)
# Morganフィンガープリントを計算
radius = 2 # Morganフィンガープリントの半径を指定
adrenaline_bit_info = {} #bitが1である情報を入れる空の辞書
dopamine_bit_info = {} #bitが1である情報を入れる空の辞書
adrenaline_fp = AllChem.GetMorganFingerprintAsBitVect(adrenaline, radius, bitInfo=adrenaline_bit_info)
dopamine_fp = AllChem.GetMorganFingerprintAsBitVect(dopamine, radius, bitInfo=dopamine_bit_info)
# アドレナリンとノンアドレナリンの共通する部分構造を取得
common_bits = [i for i, (bit1, bit2) in enumerate(zip(adrenaline_fp, dopamineb_fp)) if bit1 == 1 & bit2 == 1]
common_bit_info = {}
for i in common_bits:
list = []
for j in adrenaline_bit_info[i]:
list.append(j)
tpl = tuple(list)
common_bit_info[i]=tpl
bits_info = ((adrenaline, bit, common_bit_info) for bit in common_bit_info.keys())
# 共通する部分構造を描画
common_legend = [f'bit_No. {str(i)}' for i in common_bit_info.keys()]
Draw.DrawMorganBits(bits_info, molsPerRow=5, legends=common_legend)
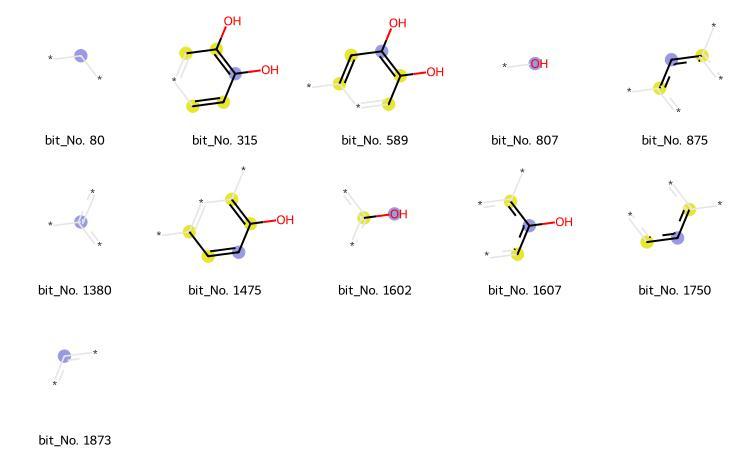
出力された共通部分構造は16個でした。
元々、アドレナリンのフィンガープリントが31個でしたので、共通する部分構造で考えると半分くらいになったということですね。
複数の化合物の共通部分構造を可視化
最後に複数の化合物の共通部分構造を可視化してみました。
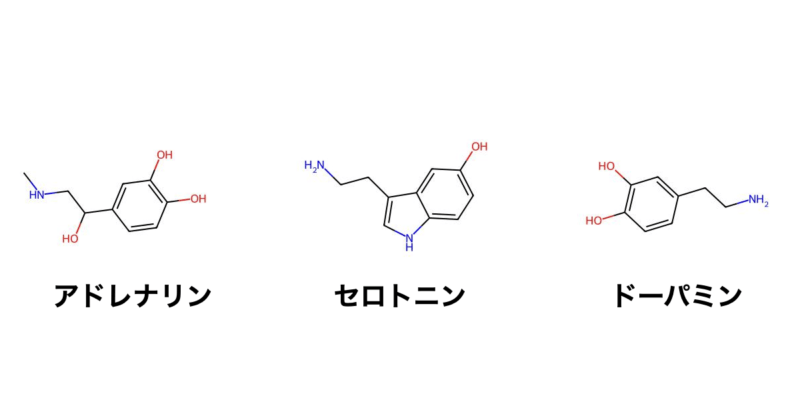
今回はアドレナリン、ドーパミン、セロトニンの3つの化合物について共通する部分構造を描画してみました。
#複数の化合物における共通部分構造を描画するプログラム
from rdkit import Chem
from rdkit.Chem import AllChem
from rdkit.Chem import Draw
#化合物の数を定義
com_num = 3
#SMILESのリストを作成
smiles_list = ["CNCC(C1=CC(=C(C=C1)O)O)O", "c1cc(c(cc1CCN)O)O", 'NCCc1c[nH]c2ccc(O)cc12']
# 空の辞書を格納するリストを作成
dict_list = []
# 空の辞書を自動で生成してリストに追加
for i in range(com_num):
dict_name = f'dict{i}'
dict_list.append({dict_name: {}})
# SMILESからMolオブジェクトを生成
mol_object_list = [] #molオブジェクトを格納する空のリスト
for smiles in smiles_list:
mol_object = Chem.MolFromSmiles(smiles)
mol_object_list.append(mol_object)
#Morganフィンガープリントを算出
radius = 2 # Morganフィンガープリントの半径を指定
for mol_object, dict_num in zip(mol_object_list, range(com_num)):
AllChem.GetMorganFingerprintAsBitVect(mol_object, radius, bitInfo=dict_list[dict_num])
# 共通する部分構造を取得
common_bits = set(dict_list[0])
for i in range(1,com_num):
common_bits = common_bits & set(dict_list[i])
common_bit_info = {}
for i in common_bits:
list = []
for j in dict_list[0][i]:
list.append(j)
tpl = tuple(list)
common_bit_info[i]=tpl
bits_info = ((mol_object_list[0], bit, common_bit_info) for bit in common_bit_info.keys())
# 共通する部分構造を描画
common_legend = [f'bit_No. {str(i)}' for i in common_bit_info.keys()]
Draw.DrawMorganBits(bits_info, molsPerRow=5, legends=common_legend)
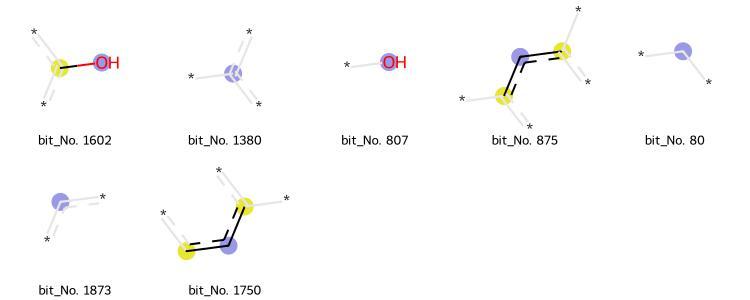
描画された共通する部分構造は7個でした。
化合物が増えていくと流石に共通する部分構造も少なくなってきますね。
ちなみにコード中のcom_numとsmiles_listを変更すればさらに多くの化合物の共通部分構造を照会することができます。
終わりに
以上がPythonを使用したフィンガープリントの部分構造の出力になります。フィンガープリントって基本的には0 or 1でしか表示されないので、視覚的に理解しにくいですが、部分構造を描画することでかなり解消された気がしますね。