こんにちは!ぼりたそです!
今回はPythonで化合物のフィンガープリントを出力する方法について解説していきます。
この記事は以下のポイントでまとめています。
- フィンガープリントとは?
- 記述子との違い
- Pythonによるフィンガープリントの出力
化学構造のフィンガープリントとは
まず化学構造におけるフィンガープリントについて説明していきます。
フィンガープリントは、分子の構造や性質をバイナリビット(0または1)や数値の形式で表現するものであり、分子構造の特定のパターンや特性を分析することができます。
主な使われ方としては、類似性評価や分子の検索、クラスタリング、機械学習などが挙げられ、分子同士の構造的な比較に役立ちます。
例えば下の図のようにアドレナリンの構造があったとします。
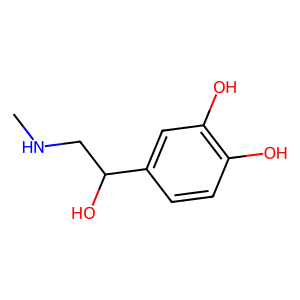
フィンガープリントに変換した場合、ベンゼン環の部分構造を含むか?というビットに対しては含むので「1」となりますが、カルボキシル基の部分構造を含むか?というビットに対しては含まないので「0」と記述されます。
このように部分構造を含むかどうかを「0」か「1」で表し、それを数百個並べることで分子構造の特徴を記述しているわけですね。
記述子との違い
次にフィンガープリントと似たパラメータとして記述子が挙げられますが、この二つの違いについて説明していきます。
記述子は、分子の性質や特性を数値や文字列で表現する方法です。これには、分子の物理的・化学的性質、立体構造、電荷、溶解度、分子量、極性などが含まれます。
記述子は、分子の特性を数値化しているため、統計的モデリング、機械学習、薬物デザインなどの分野で使用されます。
フィンガープリントと記述子の違いとして挙げられるのは以下の点です。
- 表記方法
→フィンガープリントは部分構造の有無がバイナリビット(0 or 1)で表現されるのに対して、記述子は物性値が数値として定量的に表現されています。 - 表現力
→フィンガープリントは、分子内の特定の構造的パターンを表現するのに長けているのに対して、記述子は、分子の物理的・化学的性質や特性を数値化し、表現することに長けています。 - 計算効率
→フィンガープリントは主にバイナリビットで表記されるため計算効率が高いのに対し、記述子は多くの数値または定量的な情報を提供し、より詳細な特性の評価に適しています。
Pythonによるフィンガープリントの出力
それでは実際にPythonによるフィンガープリントの出力を行なっていきます。
今回はアドレナリンの構造をフィンガープリントの一つであるMACCS keysとして出力していきたいと思います。

MACCS (Molecular Access System for Screening and Selection) keysは167個のバイナリビット(0 or 1)で表され、それぞれ特定の部分構造を含むか含まないかの二択で化学構造を表現しています。
これにより、分子間の化学的な類似性や差異を評価するのに役立ちます。例えば、特定の官能基、環構造、結合パターンなどが該当しますね。
そのため、MACCS keysは大規模な化学データベースの高速な検索やバーチャルスクリーニングに非常に有用とされています。
話を本題に戻しますが、MACSS keysを計算するために、RDKitというPythonの化学情報処理ライブラリを使用することが一般的です。
RDKitは分子情報を操作し、化学的な計算を行うための豊富なツールを提供しています。
まず、jupyter notebookなどのRDKitをインストールしていない場合は、次のコマンドを使用してインストールしてください。
また、RDKitを使用する際はjupyter notebookなどの画像や図が表示できる環境で実行するようにしましょう。
!pip install rdkit-pypi次に実際にアドレナリンのMACCS keysを計算し、データフレーム化するコードを示します。
手順としてはアドレナリンのSMILESからRDkitを使ってMACCS keysを計算して、最終的にデータフレームに格納するフローになっています。
import pandas as pd
from rdkit import Chem
from rdkit.Chem import MACCSkeys
# アドレナリンのSMILES表記
adrenaline_smiles = "CNCC(C1=CC(=C(C=C1)O)CO)O"
# 分子を生成
mol = Chem.MolFromSmiles(adrenaline_smiles)
if mol is not None:
# MACCS keysを計算
maccs_keys = MACCSkeys.GenMACCSKeys(mol)
# MACCS keysをデータフレームに変換
maccs_df = pd.DataFrame([list(maccs_keys)], columns=[f'MACCS_{i+1}' for i in range(len(maccs_keys))])
# データフレームを表示
print(maccs_df)
else:
print("Invalid SMILES")
実行してみると167個のバイナリビットが出力されるはずです。いくつかの化合物を一度に計算したい場合はリストなどにSMILESを格納して、繰り返し処理をすればいいはずです。
ついでに出力されたMACCS keysの中に0と1がそれぞれいくつ含まれるか出力してみましょう。
from rdkit import Chem
from rdkit.Chem import MACCSkeys
# アドレナリンのSMILES表記
adrenaline_smiles = "CNCC(C1=CC(=C(C=C1)O)O)O"
# SMILESから分子オブジェクトを作成
molecule = Chem.MolFromSmiles(adrenaline_smiles)
if molecule is not None:
# MACCS keysを計算
maccs_keys = MACCSkeys.GenMACCSKeys(molecule)
# MACCS keysの0と1の合計を計算
total_ones = sum(maccs_keys)
total_zeros = len(maccs_keys) - total_ones
# 合計を出力
print("Total Ones:", total_ones)
print("Total Zeros:", total_zeros)
else:
print("Invalid SMILES representation")
#出力結果
#Total Ones: 31
#Total Zeros: 1361が31個、0が136個含まれているようですね。バイナリビットと言えども167個もあれば、相当な数の化合物を表現できそうですね…
以上が化合物をフィンガープリントであるMACCS keysとして出力する方法になります。
終わりに
以上がPythonで化合物のフィンガープリントを出力する方法についての解説になります。フィンガープリントは化合物の類似性や機械学習など幅広く使用されているので、覚えておいて損はないと思います。
実はフィンガープリントにもいくつか種類があるので、後ほど比較した記事を書ければと思っています。