こんにちは!ぼりたそです!
今回はPythonライブラリであるGPyOptを用いてベイズ最適化による化合物探索プログラムを実装したので、ご紹介します。
データセットはPubChemから抽出したものを使用しています。
この記事は以下のポイントでまとめています。
- GPyOptとは?
- 探索のフロー
- 探索条件
- 使用したコード
- 探索結果
今回使用したデータを以下からダウンロードできます。data_yが目的変数、data_xが説明変数となっています。
※CSVではアクセスできないようなので、エクセルファイル(.xlsx)をダウンロードしてCSVとしてエクスポートしていただけますと使用できるかと思います。
それでは詳細に解説していきます!
GPyOptについて
まずはGPyOptについて簡単に説明します。
GPyOptは、ベイズ最適化(Bayesian Optimization)のためのPythonライブラリで、ベイズ最適化を容易に実装できるように設計されています。
主な特徴は以下の通りです。
■GPyOptの特徴
GPyOptについては以前解説した記事があるので、ご参考いただければと思います。
探索のフロー
次に今回の探索フローについて説明いたします。
探索フローとしては以下の図の通りとなります。
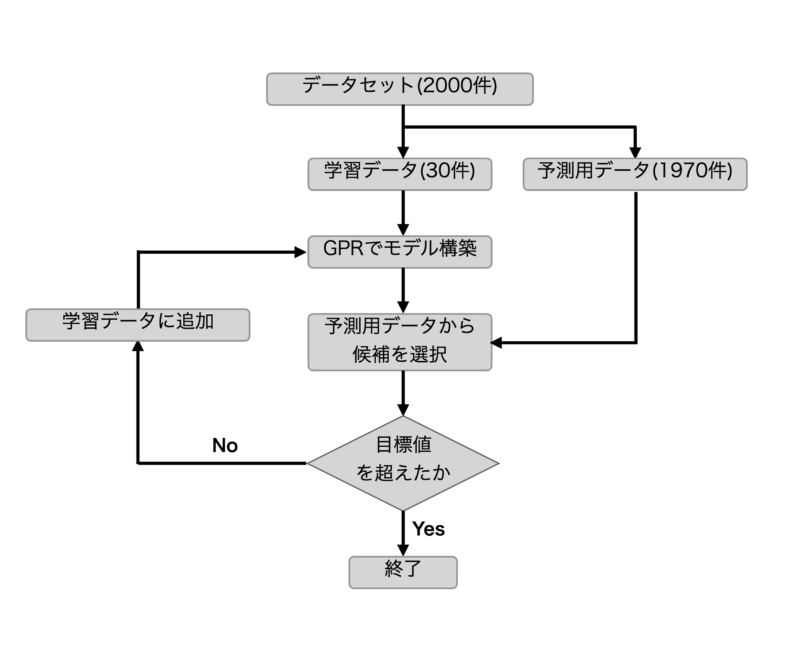

今回、用意したデータセット約2000件に対して、まずは学習データ30件、予測用データ1970件に分割します。
次に学習データを使用してGPR(ガウス過程回帰)を使用して予測モデルを構築します。
その予測モデルを使用して予測用データから次の候補を選択します。
その候補が実際に目標値を超えていれば、探索は終了とし、超えていなければ候補を新たに学習データに加え、再度予測を行うフローをループさせます。
探索条件
次に探索条件についてご説明します。
主な条件としては以下のようになっています。
| 使用ライブラリ | GPyOpt |
| 説明変数 | 化合物のMordred記述子 |
| 目的変数 | 化合物のXlogP |
| 目標値 | 予測データの最大値(XlogP=22.4) |
| 学習モデル | GPR |
| カーネル | RBF |
| 獲得関数 | EI |
今回はPubChemから抽出した化合物データを使用しております。説明変数は化合物のMordred記述を使用しています。また、目的変数は化合物のXlogP(脂溶性、水溶性の指標)を設定しています。
学習モデルはGPR(ガウス過程回帰)でカーネルはRBF(ガウシアンカーネル)となっています。獲得関数はEIを使用しています。
使用したコード
次に使用したコードについてご説明します。
コードは大まかに以下に分かれています。
- データ分割
- ベイズ最適化
それでは順に説明していきます。
データ分割
まずはデータ分割についてです。
データ分割を実行したコードは以下の通りとなります。
#データセット約2000件を学習用データ、予測用データに分割して保存。
#seed値を0〜90として10セット生成
import pandas as pd
import random
# CSVファイルの読み込み
data_x = pd.read_csv('data_x.csv', index_col='cid')
data_y = pd.read_csv('data_y.csv', index_col='cid')
# 抽出する行数が元の行数より多い場合は、元の行数に合わせる
num_rows = min(30, len(data_x))
#Seed値を設定
seed = [x for x in range(0,91) if x%10==0]
for seed in seed:
# ランダムに行を抽出
random_rows = data_x.sample(n=num_rows, random_state=random.seed(seed))
# 抽出した行を別のファイルに保存する場合
random_rows.to_csv(f'train_x_seed_{seed}.csv', index='cid')
data_y.loc[random_rows.index].to_csv(f'train_y_seed_{seed}.csv', index='cid')
# 元のデータから抽出した行を削除
data_x = data_x.drop(random_rows.index)
data_y = data_y.drop(random_rows.index)
# 更新されたデータを別のファイルに保存する場合
data_x.to_csv(f'prediction_data_x_seed_{seed}.csv', index='cid')
data_y.to_csv(f'prediction_data_y_seed_{seed}.csv', index='cid')
コードとしては、あらかじめ用意しておいたdata_y, data_xを使用して学習データ30個、予測データ1970個のデータセットを10セット生成するコードとなっております。
学習データの分割はランダムですが、シード値の設定により再現性は担保されています。
ベイズ最適化
次にベイズ最適化を実行するコードについてご説明します。
使用したコードは以下の通りです。
import pickle
import GPy
import GPyOpt
import numpy as np
import pandas as pd
from matplotlib import pyplot as plt
from sklearn.preprocessing import StandardScaler
from scipy.stats import norm
#目的関数の設定。今回は用意した候補から選択するためboundsは使用しないが、最適化が実行できないのでダミーとして設定
def f(x):
global trial
#print(f'trial:{trial}')
X_1st = x[0]
y = float(input('実験値は?'))
return y
#探索範囲の設定。今回は用意した候補から選択するためboundsは使用しないが、最適化が実行できないのでダミーとして設定
bounds = []
x.columns.tolist()
for criptor in x.columns.tolist():
bound = {}
bound['name'] = criptor
bound['type'] = 'discrete'
bound['domain'] = x_prediction[criptor].tolist()
bounds.append(bound)
#10個のデータセットについてベイズ最適化を実行
for seed in [seed for seed in range(0,91,10)]:
#試行回数を初期化
trial=1
# 予測用データをCSVファイルからデータを読み込む
x_prediction = pd.read_csv(f'prediction_data_x_seed_{seed}.csv', index_col=0, header=0)
y_answer = pd.read_csv(f'prediction_data_y_seed_{seed}.csv', index_col=0, header=0)
# 学習用の目的変数と説明変数をCSVファイルから読み込む
initial_y = pd.read_csv(f'train_y_seed_{seed}.csv', index_col=0, header=0) # 目的変数
initial_y = initial_y.iloc[:,0]
initial_y = np.array(initial_y)
initial_x = pd.read_csv(f'train_x_seed_{seed}.csv', index_col=0, header=0) # 説明変数
max_initial_y = np.max(initial_y)
suggested_y = 0
suggested_data = []
#学習データの最大値を更新するか、試行回数が200回を超えるまで繰り返しベイズ最適化を行う
while max_initial_y > float(suggested_y) and trial < 200:
print(f'試行回数:{trial}')
# 標準化のためのスケーラーを作成
scaler = StandardScaler()
#予測用データの標準化を行う
autoscaled_x_prediction = pd.DataFrame(scaler.fit_transform(x_prediction), columns=x_prediction.columns)
autoscaled_x_prediction = np.array(autoscaled_x_prediction)
x_prediction_array = np.array(x_prediction)
#目的関数の標準化を行う
initial_y = initial_y.reshape(-1,1)
mean_of_y = initial_y.mean()
std_of_y = initial_y.std()
autoscaled_initial_y = (initial_y - initial_y.mean()) / initial_y.std()
autoscaled_initial_y = np.array(autoscaled_initial_y)
autoscaled_initial_y = autoscaled_initial_y.reshape(-1,1)
# 標準化のためのスケーラーを作成
scaler = StandardScaler()
#説明変数の標準化を行う
autoscaled_initial_x = pd.DataFrame(scaler.fit_transform(initial_x), columns=initial_x.columns)
autoscaled_initial_x = np.array(autoscaled_initial_x)
# カーネルの選択
kernel_choices = [GPy.kern.RBF(input_dim=initial_x.shape[1]), GPy.kern.Matern52(input_dim=initial_x.shape[1])]
selected_kernel = kernel_choices[0] # カーネルを選択
#ベイズ最適化の条件を設定
opt = GPyOpt.methods.BayesianOptimization(f=f, domain=bounds,
model_type='GP',
#kernel=GPy.kern.RBF(input_dim=initial_x.shape[1],ARD=True),
X=autoscaled_initial_x,
Y=autoscaled_initial_y,
initial_design_numdata=len(initial_y),
maximize = True,
normalize_Y = False,
) # acquisition_weightを調整
#既にサンプリングした点を除くためにignored_Xで入力点を指定
x_suggest = opt.suggest_next_locations(ignored_X=autoscaled_initial_x)
y_predict = opt.model.model.predict(x_suggest)
#学習したGPRモデルを使用して予測
gp_model = opt.model.model
gp_mean = gp_model.predict(autoscaled_x_prediction)[0]
gp_std = gp_model.predict(autoscaled_x_prediction)[1]
current_optimum = np.max(initial_y)
#予測用データのEIを計算
EI_test = []
xi = 0.01
for i in range(len(gp_mean)):
mean = gp_mean[i]
std = gp_std[i]
f_max = np.max(initial_y)
z = (mean - f_max - xi) / std
ei = (mean - f_max - xi) * norm.cdf(z) + std * norm.pdf(z)
EI_test.append(ei)
#獲得関数をグラフ化
fig = plt.figure(figsize=(8, 6))
plt.plot(EI_test)
plt.xlabel('$x$', fontsize=16)
plt.ylabel('Expected Improvement', fontsize=16)
plt.tick_params(labelsize=16)
plt.show()
#獲得関数が最も大きかったデータを学習データに追加し、予測用データから削除する
suggested = x_prediction.iloc[EI_test.index(np.max(EI_test)),:]
initial_x = initial_x.append(suggested)
x_prediction = x_prediction.drop(index=x_prediction.index[EI_test.index(np.max(EI_test))])
suggested_y = y_answer.iloc[EI_test.index(np.max(EI_test)),:]
initial_y = np.insert(initial_y, len(initial_y), suggested_y)
initial_y = initial_y.reshape(-1,1)
y_answer = y_answer.drop(y_answer.index[EI_test.index(np.max(EI_test))])
print(f'選択されたxlogp : {suggested_y}\n'
f'トレーニングデータの最大値 : {max_initial_y}')
#推奨された候補をリストへ追加
suggested_data.append(float(suggested_y))
# 保存するリスト
sava_list = suggested_data
# リストを保存するファイルパス
file_path = f'suggested_data_seed_{seed}.pkl'
# リストを保存
with open(file_path, 'wb') as f:
pickle.dump(sava_list, f)
trial += 1このコードはGPyOptを使用してベイズ最適化を実行するコードとなります。GPyOptであれば通常、探索範囲(bounds)を設定し、その中から次の実験候補を獲得関数でスコアリングします。
しかし、今回はあらかじめ用意した化合物データから候補を選択させたいので、GPyOptでGPRのモデル作成後、予測用の化合物データから獲得関数(EI)を計算して次の候補を選択するようにしています。
探索結果
最後に実際に探索した結果について説明します。
先ほどのベイズ最適化のコードを使用した結果が以下の通りとなります。
| seed | 実探索数 |
|---|---|
| 0 | 41 |
| 10 | 39 |
| 20 | 37 |
| 30 | 36 |
| 40 | 34 |
| 50 | 33 |
| 60 | 32 |
| 70 | 36 |
| 80 | 27 |
| 90 | 32 |
| 平均 | 35 |
結果としては平均35回で目標値である予測データの最大値を引き当てていますね。1970個のデータから1つのサンプルを引き当てるのに35回なので効率はいいかと思います。
seed値で探索回数のばらつきもそこまでなく、おおよそ30〜40回で探索終了となっています。今回のデータセットでは学習データに依存せずにゴールに辿り着けていますね。
終わりに
以上がGPyOptを使用した化合物探索の実装になります。説明変数と目的変数さえ用意できれば他の実験条件などの探索にも使用できるので、参考にしていただければと思います。今後は他のベイズ最適化ライブラリでも同様の化合物探索を実装できればと思っています。