こんにちは!ぼりたそです!今回はPubChemからデータを取得する方法についてご紹介します。
最終的には2000種の有機化合物データを取得してPythonによりデータフレーム化しているので、機械学習などで使用される方はご参考までに。
この記事は以下のポイントでまとめています。
- PubChemからのデータ取得方法
- 取得データのPythonによるデータフレーム化
今回の記事では化合物データ収集について実際に行なったことをご紹介します。
化合物の検索
それではまず、PubChemから化合物を検索する方法について解説していきます。
始めにPubChemのサイトにアクセスしてください。
以下のURLからでもアクセスできます。
PubChemサイトURL↓↓↓↓↓
https://pubchem.ncbi.nlm.nih.gov/
化合物の情報から検索
PubChemにアクセスすると以下画像の赤枠のような検索窓があります。
化合物の名前や化学式からから検索したい場合は赤枠内に情報を入力してください。
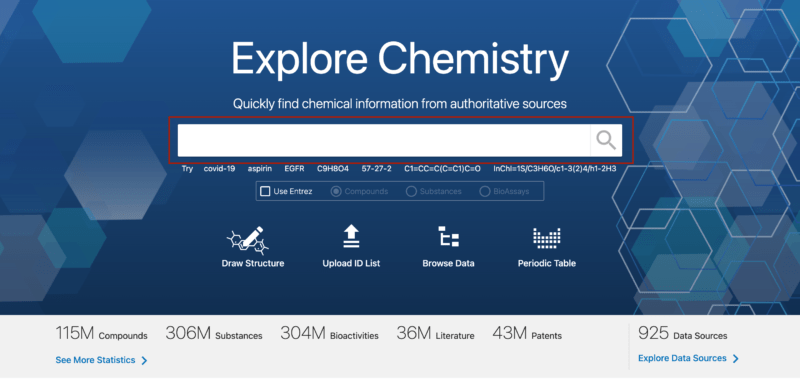
化合物の検索については、化合物名や化学式など以下の情報から検索することができます。
- 化合物名
- 化学式
- CAS No.
- CID(PubChem内のID)
- SMIELS
- InChI
部分構造から検索
一つの化合物ではなく、ある部分構造を含む化合物で検索したい場合もあるかと思います。
その場合はSMARTSを用いて検索を行います。SMARTSとはSMILESの派生みたいな表記法であり、部分構造の表現法の一つとして認識いただければと思います。
具体的な検索方法についてですが、まず下の検索窓からSMILESの式で検索してみてください。

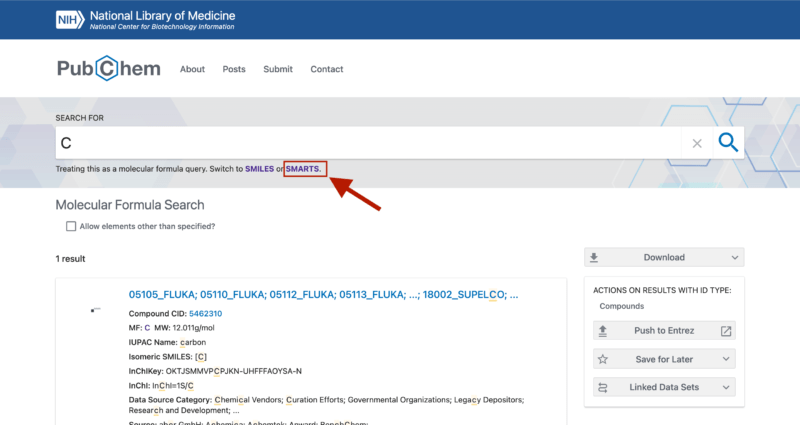
今回はC( CH4を意味する)で検索してみました。
すると以下のように化学式Cとして1件しかヒットしませんでした。
そこで画像内赤枠のSMARTSを選択します。
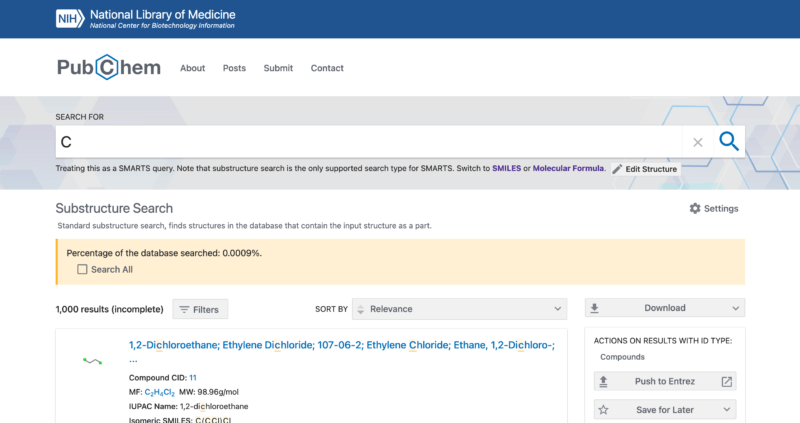
すると、以下画像の通り、Substructure Search「部分構造検索」として1000件がヒットしました。
画像のオレンジ枠の中にデータベース中の0.0009%を検索した結果とありますね。おそらく、ヒット数が多すぎるため1000件で打ち止めにされたようです。
一応、オレンジ枠の中にあるSearch Allのチェックをつければ全てのデータベースで検索してくれますが、百万件を超えるデータ数のため後でダウンロードできなくなります。
なので、ダウンロードする方はもう少し検索を絞るか、1000件で妥協しましょう。
化合物データのダウンロード
次に化合物データをダウンロード方法についてご紹介します。
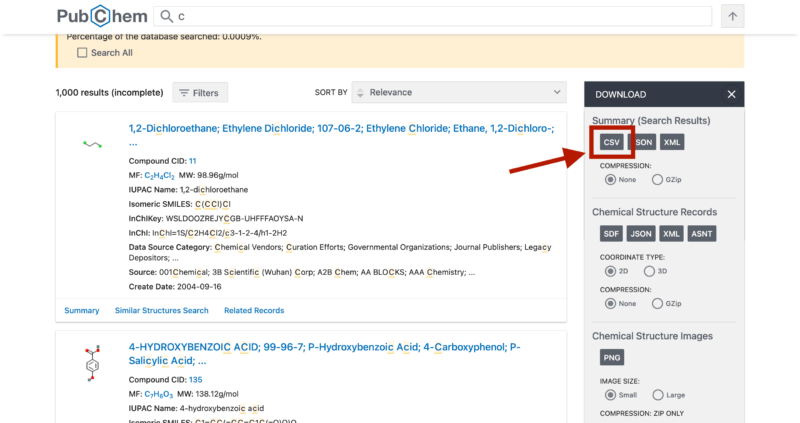
まず、検索結果の画面で以下の画像赤枠にあるようなDownloadをクリックします。
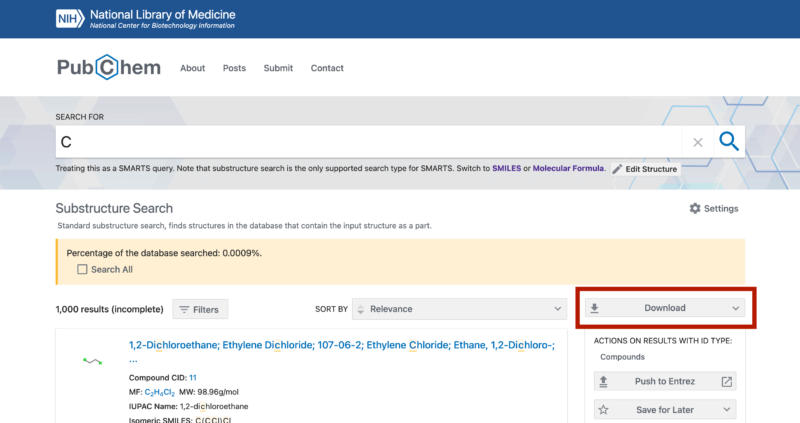
すると、以下画像のように色々オプションをつける画面が出てきますが、特に指定がない方はCSVを選択してCSVファイルとしてダウンロードすれば問題ないかと思います。
Pythonによるデータフレーム化
それではダウンロードした化合物情報をPythonによりデータフレームかする方法について紹介していきいます。
今回は以下の官能基を持つ化合物をPubChemからCSVとしてダウンロードしてデータフレーム化していきます。
- メチル基(CH3)
- ヒドロキシ基(OH)
- カルボキシル基(COOH)
- カルボニル基(C=O)
- フェニル基(C6H6)
まずは指定した官能基についてPubChemで部分構造検索を実行してそれぞれCSVファイルとしてダウンロードします。
次に以下のコードを用いてそれぞれダウンロードしたファイルを統合してデータフレーム化していきます。
※ファイル名は取得した際のデフォルトのファイル名となっています。
また、同じデータが重複している場合は削除しています。
import pandas as pd
# 結合するCSVファイルのファイル名リスト
file_names = ['PubChem_compound_smarts_C(=O).csv', 'PubChem_compound_smarts_C(=O)OH.csv', 'PubChem_compound_smarts_C1CCCCC1.csv', 'PubChem_compound_smarts_OH.csv', 'PubChem_compound_smiles_substructure_C.csv']
# 結合後のデータを格納するための空のDataFrameを作成
combined_data = pd.DataFrame()
# ファイルごとにデータを読み込んで結合する
for file in file_names:
data = pd.read_csv(file) # CSVファイルを読み込む
combined_data = combined_data.append(data, ignore_index=True) # データを結合
# 重複行を削除する
combined_data.drop_duplicates(subset=combined_data.columns[0], inplace=True)
# 結合後のデータをCSVファイルとして保存
combined_data.to_csv('combined.csv', index=False)
ちなみに、以下のコードを実行して残ったデータ数を確認してみました。
import pandas as pd
# CSVファイルを読み込む
data = pd.read_csv('combined.csv')
# カラムのデータの数をカウントする
column_counts = data['cid'].value_counts()
# 結果を表示する
print(column_counts)
#出力結果
#70 1
#342 1
#995 1
#992 1
#991 1
# ..
#456201 1
#452548 1
#449171 1
#447466 1
#12310947 1
#Name: cid, Length: 2148, dtype: int64なんと残りデータは2148個になっていました。5000個も取得したのに半分くらいは重複していたわけですね…
これは化合物の検索方法がよくなかったのかもしれませんね。汎用的な構造だったので被る化合物も多かったのかも…
まあ、それでも2000個は取得できたのでとりあえずは良しとしましょう!
今回はPubChemのwebサイトから直接データを取得しましたが、Pythonから自動で取得できる方法もあるので、知りたい方は以下の記事を参考にしていただければと思います。
終わりに
以上がPubChemから化合物データを取得した方法についての紹介でした!データ収集だけで少々味気なかったかもしれませんね。
次回は今回取得したデータについてデータ分析ができるようデータクレンジングする記事についてまとめる予定です。




