こんにちは!ぼりたそです!
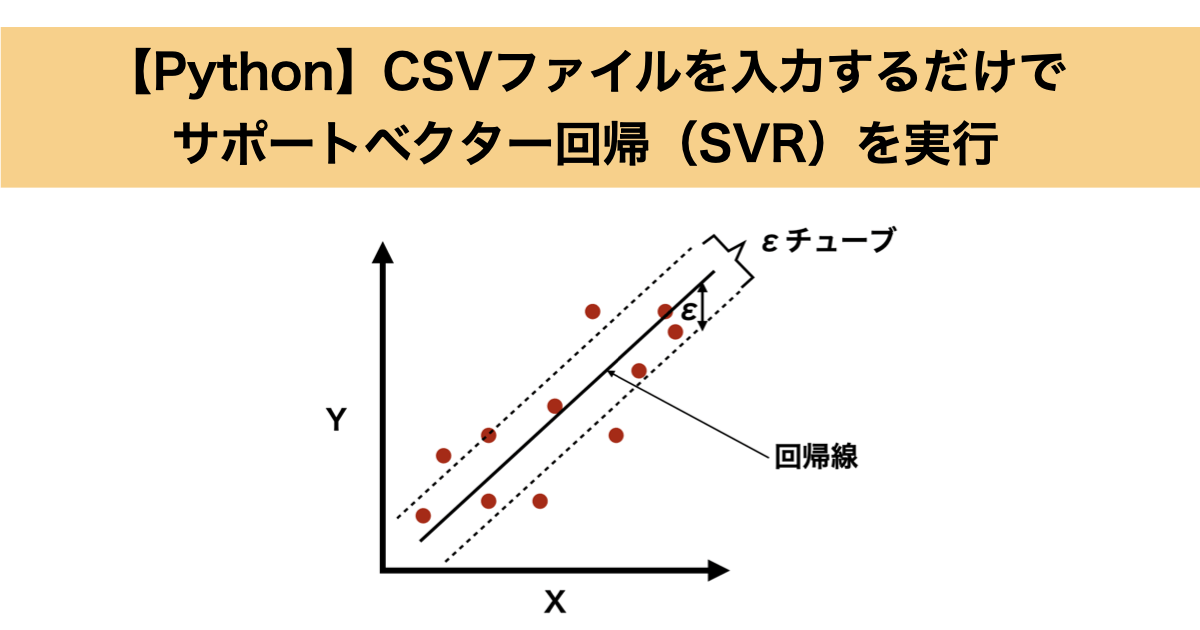
今回は以前紹介した回帰手法であるサポートベクター回帰(SVR)について、CSVファイルを入力するだけで実行できるようにPythonコードを作成しましたのでご紹介します。
この記事は以下のコンテンツから構成されています。
- サポートベクター回帰(SVR)とは?
- Pythonでの実行の流れ
- Pythonコードと解説
- 終わりに
それでは順に解説していきます。
サポートベクター回帰(SVR)とは?
サポートベクター回帰(SVR)は、サポートベクターマシン(SVM)を応用した回帰手法であり、一般の回帰手法と同様にデータに対して最適な回帰モデルを構築し、新しいデータの値を予測することが目的です。
SVMについては過去にまとめた記事がありますので、興味のある方は参照いただければと思います。
ただし、他の回帰モデルと異なるのは、「すべてのデータ点を正確に通る必要はない」という考え方です。
代わりに、一定の誤差範囲(εチューブ)内になるべくデータ点が収まるような回帰線を見つけます。これにより過学習を防ぎ、汎化性を向上することができます。
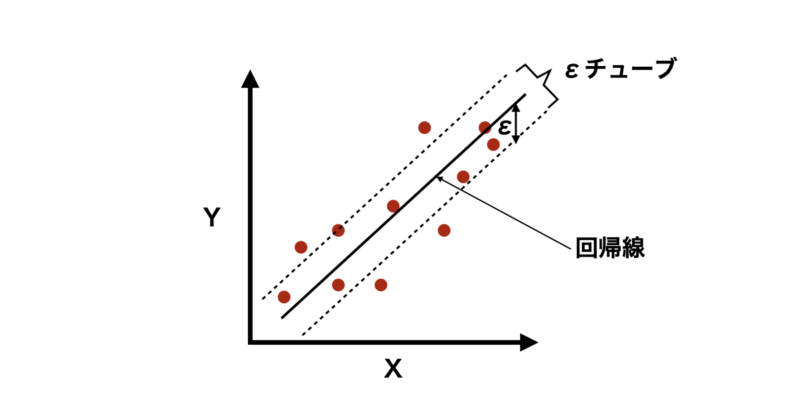

SVRの詳細については過去の記事でまとめていますので、興味のある方は参照いただければと思います。
Pythonでの実行の流れ
次に今回実装したPythonコードの実行の流れとして入力ファイルと対応する出力ファイル(アウトプット)について説明していきます。
今回のコードは学習用、予測用のCSVファイルそれぞれ入力することで解析結果や予測結果を出力するようになっています。
ファイルの詳細は以下の通りです。
◼️入力ファイル
- 学習用データ(形式:CSV)
SVRにより学習したデータをCSVにまとめたもの。一列目を目的変数、二列目以降を説明変数となるようにして下さい。また、一行目はカラム名となるようにして下さい。文字列は読み込むことができないので注意して下さい。
- 予測用データ(形式:CSV)
学習用データより構築したSVRモデルを使用して予測させるデータをCSVにまとめたもの。説明変数のみの構成とし、一列目はカラム名となるようにして下さい。文字列は読み込むことができないので注意して下さい。
◼️出力ファイル
- 説明変数の相関係数(形式:CSV)
説明変数の相関係数を計算した結果を格納したCSV。
- クロスバリデーションの結果(形式:CSV)
入力した学習データから構築したSVRモデルに対して、クロスバリデーションを実行した結果が格納されたCSV。各Foldとその平均値が格納されています。
- SHAP解析結果(形式:PNG)
入力した学習データからSHAP解析により変数重要度、beeswarm plotがグラフとして描画されたPNG。
- 予測結果(形式:CSV)
入力した予測データに対して学習済みSVRモデルを使用して予測した結果を追記したCSV。
以上が入力ファイルおよび出力ファイルに関する説明となります。
実装コードと解説
それでは実装したPythonコードと、その解説をしたいと思います。
今回実装したコードは以下の通りとなります。
import pandas as pd
import numpy as np
from sklearn.svm import SVR
from sklearn.model_selection import cross_val_score, KFold, LeaveOneOut
from sklearn.preprocessing import StandardScaler
from sklearn.metrics import r2_score
import matplotlib.pyplot as plt
from skopt import BayesSearchCV # ベイズ最適化を使用するためのライブラリ
import shap # SHAP値を計算するためのライブラリ
import sys
# CSVファイルの読み込み
def load_csv(file_path):
"""
指定されたCSVファイルを読み込み、説明変数と目的変数を分離する。
Args:
file_path (str): CSVファイルのパス
Returns:
X (numpy.ndarray): 説明変数のデータ
y (numpy.ndarray): 目的変数のデータ
data (pandas.DataFrame): 読み込んだデータ全体
feature_names (pandas.Index): 説明変数のカラム名
"""
data = pd.read_csv(file_path)
y = data.iloc[:, 0].values # 目的変数 (最初の列)
X = data.iloc[:, 1:].values # 説明変数 (2列目以降)
feature_names = data.columns[1:] # 説明変数のカラム名
return X, y, data, feature_names
# 説明変数の相関係数を計算して保存
def calculate_and_save_correlations(data, output_file):
"""
説明変数間の相関係数を計算し、CSVファイルに保存する。
Args:
data (pandas.DataFrame): データ全体
output_file (str): 出力先のCSVファイル名
"""
correlations = data.iloc[:, 1:].corr() # 説明変数の相関係数を計算
correlations.to_csv(output_file) # 結果をCSVに保存
print(f"相関係数を{output_file}に保存しました。")
# SHAP値の計算とプロット
def explain_with_shap(model, X, feature_names):
"""
SHAP値を計算し、特徴量の重要度を可視化するプロットを生成。
Args:
model: 学習済みモデル
X (numpy.ndarray): 標準化済みの説明変数データ
feature_names (pandas.Index): 説明変数のカラム名
"""
explainer = shap.KernelExplainer(model.predict, X) # SHAPの説明モデル
shap_values = explainer.shap_values(X) # SHAP値を計算
# 特徴量の重要度を棒グラフで表示
plt.figure()
shap.summary_plot(shap_values, X, feature_names=feature_names, plot_type="bar", show=False)
plt.savefig("shap_summary_bar.png")
print("SHAP Summary Plot (Bar)を保存しました: shap_summary_bar.png")
# 特徴量の分布と影響をビーズウォームプロットで表示
plt.figure()
shap.summary_plot(shap_values, X, feature_names=feature_names, show=False)
plt.savefig("shap_beeswarm.png")
print("SHAP Beeswarm Plotを保存しました: shap_beeswarm.png")
# 実測値と予測値のプロット
def plot_predictions(model, X, y, cv):
"""
クロスバリデーションを通じて得られた実測値と予測値をプロット。
Args:
model: 学習済みモデル
X (numpy.ndarray): 標準化済みの説明変数データ
y (numpy.ndarray): 実測値
cv: クロスバリデーションの分割オブジェクト
"""
predictions = []
true_values = []
# 各分割でモデルを訓練し、予測を収集
for train_idx, test_idx in cv.split(X):
model.fit(X[train_idx], y[train_idx]) # モデルを学習
y_pred = model.predict(X[test_idx]) # テストデータを予測
predictions.extend(y_pred)
true_values.extend(y[test_idx])
# 実測値 vs 予測値を散布図で表示
plt.figure(figsize=(8, 8))
plt.scatter(true_values, predictions, label="Predictions")
plt.plot([min(true_values), max(true_values)], [min(true_values), max(true_values)], 'r--', label="y=x")
plt.xlabel("True Values")
plt.ylabel("Predicted Values")
plt.title("True vs Predicted Values")
plt.legend()
plt.grid()
plt.show()
# 予測用CSVの読み込みと予測結果の保存
def predict_and_save(model, scaler, prediction_csv):
"""
指定されたCSVファイルに基づいて予測を行い、結果を新しいCSVに保存。
Args:
model: 学習済みモデル
scaler: 標準化オブジェクト
prediction_csv (str): 予測用データのCSVファイルパス
"""
predict_data = pd.read_csv(prediction_csv)
X_predict = predict_data.values # 説明変数データを取得
X_predict_scaled = scaler.transform(X_predict) # データを標準化
predictions = model.predict(X_predict_scaled) # 予測
predict_data.insert(0, "Predicted", predictions) # 予測結果をデータに追加
output_file = "predictions_output.csv"
predict_data.to_csv(output_file, index=False) # 結果を保存
print(f"予測結果を{output_file}に保存しました。")
# SVRモデルの最適化とクロスバリデーション
def train_and_evaluate_svr(X, y, feature_names, kernel='rbf', output_correlation_file='correlations.csv', output_cv_file='cv_results.csv', prediction_csv=None):
"""
SVRモデルのハイパーパラメータを最適化し、クロスバリデーションを実行。
必要に応じてSHAP値の計算や予測結果の保存も行う。
Args:
X (numpy.ndarray): 説明変数のデータ
y (numpy.ndarray): 目的変数のデータ
feature_names (pandas.Index): 説明変数のカラム名
kernel (str): SVRのカーネルタイプ
output_correlation_file (str): 相関係数の出力ファイル名
output_cv_file (str): クロスバリデーション結果の出力ファイル名
prediction_csv (str): 予測用CSVファイルのパス (省略可能)
"""
# データの標準化 (平均0, 分散1にスケーリング)
scaler = StandardScaler()
X_scaled = scaler.fit_transform(X)
# SVRのハイパーパラメータをベイズ最適化で調整
param_space = {
'C': (0.1, 100.0, 'log-uniform'), # 正則化パラメータ
'gamma': (1e-4, 1e-1, 'log-uniform'), # RBFカーネルの幅
'epsilon': (0.01, 1.0, 'uniform') # 許容誤差
}
svr = SVR(kernel=kernel)
bayes_search = BayesSearchCV(svr, param_space, n_iter=50, cv=5, scoring='r2', random_state=42)
bayes_search.fit(X_scaled, y)
best_model = bayes_search.best_estimator_ # 最適なモデル
print(f"最適なパラメータ: {bayes_search.best_params_}")
# クロスバリデーションの設定
n_samples = len(y)
if n_samples <= 30: # サンプル数が少ない場合はLeave-One-Outを使用
cv = LeaveOneOut()
else: # サンプル数が多い場合はK-Foldを使用
cv = KFold(n_splits=10, shuffle=True, random_state=42)
# クロスバリデーションでR2スコアを計算
r2_scores = cross_val_score(best_model, X_scaled, y, cv=cv, scoring='r2')
print(f"各foldのR2値: {r2_scores}")
# 結果をCSVに保存
cv_results = pd.DataFrame({
'Fold': range(1, len(r2_scores) + 1),
'R2': r2_scores
})
cv_results.loc['Mean'] = ['Mean', r2_scores.mean()] # 平均スコアを追加
cv_results.to_csv(output_cv_file, index=False)
print(f"クロスバリデーション結果を{output_cv_file}に保存しました。")
# SHAP値を計算してプロット
explain_with_shap(best_model, X_scaled, feature_names)
# 実測値と予測値のプロット
plot_predictions(best_model, X_scaled, y, cv)
# 予測CSVファイルが指定されている場合、予測結果を保存
if prediction_csv:
predict_and_save(best_model, scaler, prediction_csv)
if __name__ == "__main__":
# 学習用データと予測用データのCSVファイルパスを指定
input_csv = "train_data.csv"
prediction_csv = "prediction_data.csv"
# CSVデータの読み込み
X, y, data, feature_names = load_csv(input_csv)
# 相関係数を計算して保存
calculate_and_save_correlations(data, "correlations.csv")
# SVRモデルの訓練と評価
train_and_evaluate_svr(X, y, feature_names, kernel='rbf', output_correlation_file="correlations.csv", output_cv_file="cv_results.csv", prediction_csv=prediction_csv)
このコードの構成としては以下の通りとなります。
- ライブラリインポート
- 関数_CSVファイル読み込み
- 関数_説明変数の相関係数計算
- 関数_SHAP値の計算
- 関数_CVの予測値VS実測値プロット
- 関数_予測用CSVの読み込みと計算
- 関数_SVRのモデル構築
- SVRの実行
それぞれのブロックに分けて順に説明していきます。
単純にSVRの実行だけしたいという方は「SVRの実行」のみ参照いただければと思います。
ライブラリのインポート
まずはライブラリのインポートについてです。
実装コードでは以下の部分で処理しています。
import pandas as pd
import numpy as np
from sklearn.svm import SVR
from sklearn.model_selection import cross_val_score, KFold, LeaveOneOut
from sklearn.preprocessing import StandardScaler
from sklearn.metrics import r2_score
import matplotlib.pyplot as plt
from skopt import BayesSearchCV # ベイズ最適化を使用するためのライブラリ
import shap # SHAP値を計算するためのライブラリ
import sys本コードで必要なライブラリをインポートしています。特に説明することはありませんが、インストールしていないライブラリがあれば、「pip install ライブラリ名」で実行しインストールいただければと思います。
関数_CSVファイル読み込み
次にCSVファイルを読み込む関数について説明します。
実装コードでは以下の部分で定義しています。
# CSVファイルの読み込み
def load_csv(file_path):
"""
指定されたCSVファイルを読み込み、説明変数と目的変数を分離する。
Args:
file_path (str): CSVファイルのパス
Returns:
X (numpy.ndarray): 説明変数のデータ
y (numpy.ndarray): 目的変数のデータ
data (pandas.DataFrame): 読み込んだデータ全体
feature_names (pandas.Index): 説明変数のカラム名
"""
data = pd.read_csv(file_path)
y = data.iloc[:, 0].values # 目的変数 (最初の列)
X = data.iloc[:, 1:].values # 説明変数 (2列目以降)
feature_names = data.columns[1:] # 説明変数のカラム名
return X, y, data, feature_names関数の引数とその説明については以下の通りです。
| 引数 | 説明 |
|---|---|
| file_path | 入力したいCSVのファイルパス |
処理としては入力したCSVファイルを目的変数と説明変数に分けるのがメインとなります。また、後ほど使用するため説明変数のカラム名も抽出しています。
最終的に目的変数、説明変数、全体データ(目的変数+説明変数)、カラム名を返り値としています。
関数_説明変数の相関係数計算
次に説明変数の相関係数を計算する関数についての説明です。
実装コードでは以下の部分で実装しています。
# 説明変数の相関係数を計算して保存
def calculate_and_save_correlations(data, output_file):
"""
説明変数間の相関係数を計算し、CSVファイルに保存する。
Args:
data (pandas.DataFrame): データ全体
output_file (str): 出力先のCSVファイル名
"""
correlations = data.iloc[:, 1:].corr() # 説明変数の相関係数を計算
correlations.to_csv(output_file) # 結果をCSVに保存
print(f"相関係数を{output_file}に保存しました。")引数の説明は以下のテーブルの通りとなっています。
| 引数 | 説明 |
|---|---|
| data | 全体データ(目的変数+説明変数) |
| output_file | 相関係数が格納されたCSVの出力先のパス |
処理としては全体データから説明変数のカラムのみを抽出して説明変数同士の相関係数計算しています。
最終的に引数として設定したoutput_fileのパスに相関係数が格納されたCSVファイルが出力されます。
関数_SHAP値の計算
次にSHAP値の計算を実行する関数についての説明です。
SHAPについては過去の記事でまとめていますので、興味のある方は参考にしていただければと思います。
実装コードの中では以下の部分で処理しています。
# SHAP値の計算とプロット
def explain_with_shap(model, X, feature_names):
"""
SHAP値を計算し、特徴量の重要度を可視化するプロットを生成。
Args:
model: 学習済みモデル
X (numpy.ndarray): 標準化済みの説明変数データ
feature_names (pandas.Index): 説明変数のカラム名
"""
explainer = shap.KernelExplainer(model.predict, X) # SHAPの説明モデル
shap_values = explainer.shap_values(X) # SHAP値を計算
# 特徴量の重要度を棒グラフで表示
plt.figure()
shap.summary_plot(shap_values, X, feature_names=feature_names, plot_type="bar", show=False)
plt.savefig("shap_summary_bar.png")
print("SHAP Summary Plot (Bar)を保存しました: shap_summary_bar.png")
# 特徴量の分布と影響をビーズウォームプロットで表示
plt.figure()
shap.summary_plot(shap_values, X, feature_names=feature_names, show=False)
plt.savefig("shap_beeswarm.png")
print("SHAP Beeswarm Plotを保存しました: shap_beeswarm.png")関数の引数とその説明は以下のテーブルの通りです。
| 引数 | 説明 |
|---|---|
| model | 学習済みモデル |
| X | 標準化済みの説明変数 |
| feature_name | 説明変数の名称 |
処理としては、まず学習済みモデルを使用してSHAP値を計算しています。この時ExplainerはKernelExplainerを使用しています。
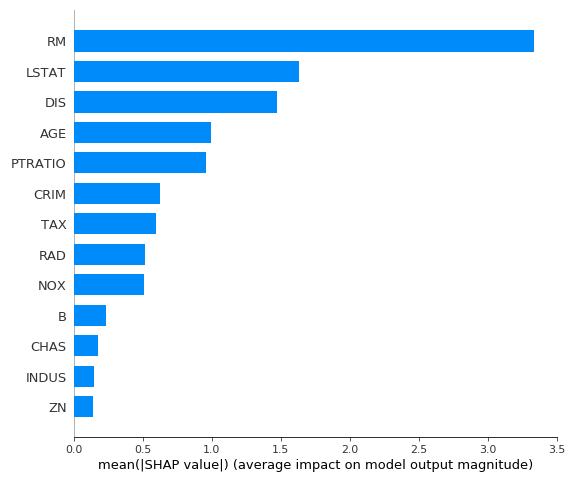
次に計算したSHAP値から説明変数の重要度を棒グラフとして出力しています。出力形式はpngでファイル名はshap_summary_bar.pngとして出力されます。
例として、ボストン住宅価格のデータを入力すると以下のような結果が出力されています。

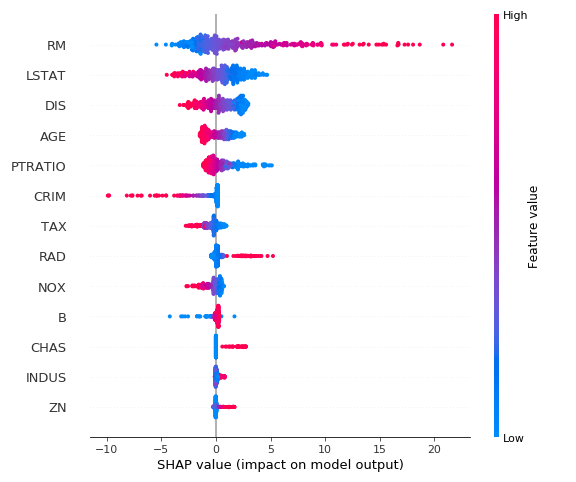
さらに、説明変数の分布と影響を可視化するbeeswarmplotを出力します。これも出力形式はpngでファイル名はshap_beeswarm.pngとして出力されます。
こちらも例としてボストン住宅価格のデータを入力すると、以下のような結果が出力されました。

関数_CVの予測値VS実測値プロット
次にCV(クロスバリデーション)から得られる予想値VS実測値プロットを出力する関数についての説明です。
CVについては過去に記事をまとめましたので、興味のある方は参照いただければと思います。
実装コードでは以下の部分で処理しています。
# 実測値と予測値のプロット
def plot_predictions(model, X, y, cv):
"""
クロスバリデーションを通じて得られた実測値と予測値をプロット。
Args:
model: 学習済みモデル
X (numpy.ndarray): 標準化済みの説明変数データ
y (numpy.ndarray): 実測値
cv: クロスバリデーションの分割オブジェクト
"""
predictions = []
true_values = []
# 各分割でモデルを訓練し、予測を収集
for train_idx, test_idx in cv.split(X):
model.fit(X[train_idx], y[train_idx]) # モデルを学習
y_pred = model.predict(X[test_idx]) # テストデータを予測
predictions.extend(y_pred)
true_values.extend(y[test_idx])
# 実測値 vs 予測値を散布図で表示
plt.figure(figsize=(8, 8))
plt.scatter(true_values, predictions, label="Predictions")
plt.plot([min(true_values), max(true_values)], [min(true_values), max(true_values)], 'r--', label="y=x")
plt.xlabel("True Values")
plt.ylabel("Predicted Values")
plt.title("True vs Predicted Values")
plt.legend()
plt.grid()
plt.show()関数の引数とその説明は以下の通りです。
| 引数 | 説明 |
|---|---|
| model | 学習済みモデル |
| X | 説明変数 |
| y | 目的変数 |
| cv | CVの分割オブジェクト(LeaveOneOut, KFoldなど) |
処理としては、引数として入力したcv分割オブジェクト(cv)に従って説明変数、目的変数を分割します。その後、実際にCVを実行して、実測値と予測値を散布図としてグラフ化します。
精度が良いモデルであれば予測値と実測値が一直線に並ぶようなプロットになるはずです。
関数_予測用CSVの読み込みと計算
次に予測用CSVの読み込みと計算を実行する関数について説明します。
実装コードの中で以下の部分で処理しています。
# 予測用CSVの読み込みと予測結果の保存
def predict_and_save(model, scaler, prediction_csv):
"""
指定されたCSVファイルに基づいて予測を行い、結果を新しいCSVに保存。
Args:
model: 学習済みモデル
scaler: 標準化オブジェクト
prediction_csv (str): 予測用データのCSVファイルパス
"""
predict_data = pd.read_csv(prediction_csv)
X_predict = predict_data.values # 説明変数データを取得
X_predict_scaled = scaler.transform(X_predict) # データを標準化
predictions = model.predict(X_predict_scaled) # 予測
predict_data.insert(0, "Predicted", predictions) # 予測結果をデータに追加
output_file = "predictions_output.csv"
predict_data.to_csv(output_file, index=False) # 結果を保存
print(f"予測結果を{output_file}に保存しました。")関数の引数とその説明は以下の通りです。
| 引数 | 説明 |
|---|---|
| model | 学習済みモデル |
| scaler | 標準化、正規化などのスケーリングオブジェクト |
| prediction_csv | 予測用CSVのファイルパス |
処理としては、まず予測用CSVのパスから説明変数を読み込み、スケーリングオブジェクトで正規化または標準化を実行します。
次に学習済みモデルを使用して標準化した説明変数から目的変数の予測を行います。
最後に予測した目的変数を予測用CSVに追加してoutput_fileとしてCSVファイルで出力されます。
関数_SVRのモデル構築
次にSVRのモデル構築を実行する関数について説明します。
実装コードの中ではこの関数がメインとなっており、これまで説明してきた関数を使用してSVRのモデル構築やモデル精度の検証などを行います。
実装コードの中では以下の部分で処理しています。
# SVRモデルの最適化とクロスバリデーション
def train_and_evaluate_svr(X, y, feature_names, kernel='rbf', output_correlation_file='correlations.csv', output_cv_file='cv_results.csv', prediction_csv=None):
"""
SVRモデルのハイパーパラメータを最適化し、クロスバリデーションを実行。
必要に応じてSHAP値の計算や予測結果の保存も行う。
Args:
X (numpy.ndarray): 説明変数のデータ
y (numpy.ndarray): 目的変数のデータ
feature_names (pandas.Index): 説明変数のカラム名
kernel (str): SVRのカーネルタイプ
output_correlation_file (str): 相関係数の出力ファイル名
output_cv_file (str): クロスバリデーション結果の出力ファイル名
prediction_csv (str): 予測用CSVファイルのパス (省略可能)
"""
# データの標準化 (平均0, 分散1にスケーリング)
scaler = StandardScaler()
X_scaled = scaler.fit_transform(X)
# SVRのハイパーパラメータをベイズ最適化で調整
param_space = {
'C': (0.1, 100.0, 'log-uniform'), # 正則化パラメータ
'gamma': (1e-4, 1e-1, 'log-uniform'), # RBFカーネルの幅
'epsilon': (0.01, 1.0, 'uniform') # 許容誤差
}
svr = SVR(kernel=kernel)
bayes_search = BayesSearchCV(svr, param_space, n_iter=50, cv=5, scoring='r2', random_state=42)
bayes_search.fit(X_scaled, y)
best_model = bayes_search.best_estimator_ # 最適なモデル
print(f"最適なパラメータ: {bayes_search.best_params_}")
# クロスバリデーションの設定
n_samples = len(y)
if n_samples <= 30: # サンプル数が少ない場合はLeave-One-Outを使用
cv = LeaveOneOut()
else: # サンプル数が多い場合はK-Foldを使用
cv = KFold(n_splits=10, shuffle=True, random_state=42)
# クロスバリデーションでR2スコアを計算
r2_scores = cross_val_score(best_model, X_scaled, y, cv=cv, scoring='r2')
print(f"各foldのR2値: {r2_scores}")
# 結果をCSVに保存
cv_results = pd.DataFrame({
'Fold': range(1, len(r2_scores) + 1),
'R2': r2_scores
})
cv_results.loc['Mean'] = ['Mean', r2_scores.mean()] # 平均スコアを追加
cv_results.to_csv(output_cv_file, index=False)
print(f"クロスバリデーション結果を{output_cv_file}に保存しました。")
# SHAP値を計算してプロット
explain_with_shap(best_model, X_scaled, feature_names)
# 実測値と予測値のプロット
plot_predictions(best_model, X_scaled, y, cv)
# 予測CSVファイルが指定されている場合、予測結果を保存
if prediction_csv:
predict_and_save(best_model, scaler, prediction_csv) 関数の引数とその説明は以下の通りです。
| 引数 | 説明 |
|---|---|
| X | 説明変数 |
| y | 目的変数 |
| feature_names | 説明変数のカラム名(グラフや出力CSVに使用) |
| kernel | SVRで使用するカーネル。デフォルトはRBFで設定 |
| output_correlation_file | 説明変数の相関係数を出力するCSVのファイルパス |
| output_cv_file | SVRのモデル精度をCVで評価した結果を出力するCSVのファイルパス |
| prediction_csv | 予測用CSVのファイルパス。デフォルトはNoneで設定。 |
処理としては、まず読み込んだ説明変数を標準化します。
次にSVRのハイパーパラメータをベイズ最適化を用いて最適化していきます。
最適化したパラメータからSVRモデルを構築し、CVによりモデル検証を行います。CVの結果についてはoutput_cv_fileのファイルパスに従って出力されます。
次にexplain_with_shapとして定義した関数を使用して、構築したSVRモデルから変数重要度や分布などをグラフ化して出力します。
更にplot_predictionsとして定義した関数を使用して、CVで検証した実測値と予測値をグラフ化して出力します。
最後に予測用CSVが指定されていればpredict_and_saveとして定義した関数を使用して、予測した結果を再度CSVファイルとして出力します。
SVRの実行
最後にSVRを実行するコードについて説明します。
これまで定義した関数を使用して実際にSVRを実行するコードとなっています。
実装コードでは以下の部分で処理しています。
if __name__ == "__main__":
# 学習用データと予測用データのCSVファイルパスを指定
input_csv = "train_data.csv"
prediction_csv = "prediction_data.csv"
# CSVデータの読み込み
X, y, data, feature_names = load_csv(input_csv)
# 相関係数を計算して保存
calculate_and_save_correlations(data, "correlations.csv")
# SVRモデルの訓練と評価
train_and_evaluate_svr(X, y, feature_names, kernel='rbf', output_correlation_file="correlations.csv", output_cv_file="cv_results.csv", prediction_csv=prediction_csv)処理として、if name == “main“としてあるのはモジュール読み込み用として関数を読み込みたい時に実行されないようにするためです。
まず、学習用データと予測用データのCSVファイルパスを指定して読み込みます。この時、予測用CSVがない場合はNoneとして下さい。
次に読み込んだCSVファイルから説明変数、目的変数、変数名などを抽出します。その後、calculate_and_save_correlationsの関数を使用して説明変数の相関係数を計算し、結果をCSVファイルとして出力します。
最後にtrain_and_evaluate_svrの関数を使用してSVRのモデル構築および評価を行い、予測用CSVが指定していれば予測した結果を返してくれます。また、CVの結果やSHAPの結果も別ファイルで保存して出力してくれます。
以上がSVRを実行するPythonコードの説明になります。
終わりに
いかがだったでしょうか?SVRの実行だけではなく、モデル精度の検証結果や変数重要度などを出力するようにしたので、モデルの解釈性も高いのではないでしょうか。また、予測用CSVを入力しておけば、構築したモデルから予測結果を返してくれるので便利かなと思います。